注意
前往結尾 下載完整的範例程式碼。或透過 JupyterLite 或 Binder 在您的瀏覽器中執行此範例
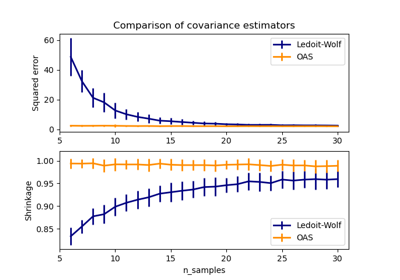
收縮共變異數估計:LedoitWolf vs OAS 和最大概似#
在使用共變異數估計時,通常的方法是使用最大概似估計器,例如 EmpiricalCovariance。它是無偏的,也就是說,當給出許多觀察值時,它會收斂到真實(母體)共變異數。然而,為了減少其變異數,對其進行正規化也可能是有益的;反過來,這會引入一些偏差。此範例說明 收縮共變異數估計器中使用的簡單正規化。特別是,它著重於如何設定正規化的量,即如何選擇偏差-變異數權衡。
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
產生樣本數據#
import numpy as np
n_features, n_samples = 40, 20
np.random.seed(42)
base_X_train = np.random.normal(size=(n_samples, n_features))
base_X_test = np.random.normal(size=(n_samples, n_features))
# Color samples
coloring_matrix = np.random.normal(size=(n_features, n_features))
X_train = np.dot(base_X_train, coloring_matrix)
X_test = np.dot(base_X_test, coloring_matrix)
計算測試資料上的概似#
from scipy import linalg
from sklearn.covariance import ShrunkCovariance, empirical_covariance, log_likelihood
# spanning a range of possible shrinkage coefficient values
shrinkages = np.logspace(-2, 0, 30)
negative_logliks = [
-ShrunkCovariance(shrinkage=s).fit(X_train).score(X_test) for s in shrinkages
]
# under the ground-truth model, which we would not have access to in real
# settings
real_cov = np.dot(coloring_matrix.T, coloring_matrix)
emp_cov = empirical_covariance(X_train)
loglik_real = -log_likelihood(emp_cov, linalg.inv(real_cov))
比較設定正規化參數的不同方法#
在這裡我們比較 3 種方法
透過根據潛在收縮參數的網格,交叉驗證三個折疊上的概似來設定參數。
Ledoit 和 Wolf 提出的緊密公式,用於計算漸近最佳正規化參數(最小化 MSE 標準),產生
LedoitWolf共變異數估計。Ledoit-Wolf 收縮的改進,由 Chen 等人提出的
OAS。在資料為高斯分佈的假設下,尤其是在小樣本的情況下,其收斂性明顯更好。
from sklearn.covariance import OAS, LedoitWolf
from sklearn.model_selection import GridSearchCV
# GridSearch for an optimal shrinkage coefficient
tuned_parameters = [{"shrinkage": shrinkages}]
cv = GridSearchCV(ShrunkCovariance(), tuned_parameters)
cv.fit(X_train)
# Ledoit-Wolf optimal shrinkage coefficient estimate
lw = LedoitWolf()
loglik_lw = lw.fit(X_train).score(X_test)
# OAS coefficient estimate
oa = OAS()
loglik_oa = oa.fit(X_train).score(X_test)
繪製結果#
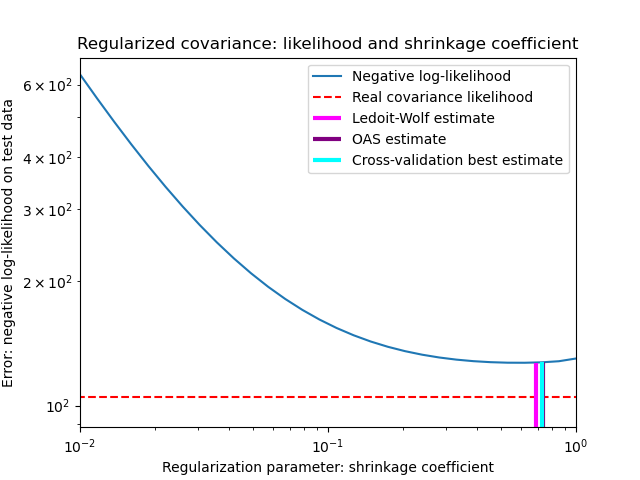
為了量化估計誤差,我們繪製了不同收縮參數值的未見資料的概似。我們還展示了透過交叉驗證,或使用 LedoitWolf 和 OAS 估計的選擇。
import matplotlib.pyplot as plt
fig = plt.figure()
plt.title("Regularized covariance: likelihood and shrinkage coefficient")
plt.xlabel("Regularization parameter: shrinkage coefficient")
plt.ylabel("Error: negative log-likelihood on test data")
# range shrinkage curve
plt.loglog(shrinkages, negative_logliks, label="Negative log-likelihood")
plt.plot(plt.xlim(), 2 * [loglik_real], "--r", label="Real covariance likelihood")
# adjust view
lik_max = np.amax(negative_logliks)
lik_min = np.amin(negative_logliks)
ymin = lik_min - 6.0 * np.log((plt.ylim()[1] - plt.ylim()[0]))
ymax = lik_max + 10.0 * np.log(lik_max - lik_min)
xmin = shrinkages[0]
xmax = shrinkages[-1]
# LW likelihood
plt.vlines(
lw.shrinkage_,
ymin,
-loglik_lw,
color="magenta",
linewidth=3,
label="Ledoit-Wolf estimate",
)
# OAS likelihood
plt.vlines(
oa.shrinkage_, ymin, -loglik_oa, color="purple", linewidth=3, label="OAS estimate"
)
# best CV estimator likelihood
plt.vlines(
cv.best_estimator_.shrinkage,
ymin,
-cv.best_estimator_.score(X_test),
color="cyan",
linewidth=3,
label="Cross-validation best estimate",
)
plt.ylim(ymin, ymax)
plt.xlim(xmin, xmax)
plt.legend()
plt.show()
注意
最大概似估計對應於沒有收縮,因此表現不佳。Ledoit-Wolf 估計表現得非常好,因為它接近最佳值,而且計算成本不高。在此範例中,OAS 估計稍微遠一些。有趣的是,這兩種方法都優於交叉驗證,後者的計算成本明顯最高。
腳本的總執行時間:(0 分鐘 0.434 秒)
相關範例