注意
前往末尾下載完整範例程式碼。或透過 JupyterLite 或 Binder 在您的瀏覽器中執行此範例
離群值偵測估算器的評估#
此範例比較兩種離群值偵測演算法,即局部離群值因子 (LOF) 和隔離森林 (IForest),在 sklearn.datasets 中可用的真實世界資料集上。目的是顯示不同的演算法在不同的資料集上表現良好,並對比它們的訓練速度和對超參數的敏感度。
演算法在假設包含離群值的整個資料集上進行訓練 (沒有標籤)。
1. ROC 曲線是使用地面真值標籤的知識計算出來的,並使用 RocCurveDisplay 顯示。
效能以 ROC-AUC 來評估。
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
資料集預處理和模型訓練#
不同的離群值偵測模型需要不同的預處理。在存在類別變數的情況下,OrdinalEncoder 對於基於樹的模型 (例如 IsolationForest) 通常是一種很好的策略,而基於鄰居的模型 (例如 LocalOutlierFactor) 將受到序數編碼引起的排序的影響。為了避免產生排序,應該使用 OneHotEncoder。
基於鄰居的模型也可能需要縮放數值特徵 (請參閱例如 重新調整對 k 近鄰模型的影響)。在存在離群值的情況下,一個好的選擇是使用 RobustScaler。
from sklearn.compose import ColumnTransformer
from sklearn.ensemble import IsolationForest
from sklearn.neighbors import LocalOutlierFactor
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import (
OneHotEncoder,
OrdinalEncoder,
RobustScaler,
)
def make_estimator(name, categorical_columns=None, iforest_kw=None, lof_kw=None):
"""Create an outlier detection estimator based on its name."""
if name == "LOF":
outlier_detector = LocalOutlierFactor(**(lof_kw or {}))
if categorical_columns is None:
preprocessor = RobustScaler()
else:
preprocessor = ColumnTransformer(
transformers=[("categorical", OneHotEncoder(), categorical_columns)],
remainder=RobustScaler(),
)
else: # name == "IForest"
outlier_detector = IsolationForest(**(iforest_kw or {}))
if categorical_columns is None:
preprocessor = None
else:
ordinal_encoder = OrdinalEncoder(
handle_unknown="use_encoded_value", unknown_value=-1
)
preprocessor = ColumnTransformer(
transformers=[
("categorical", ordinal_encoder, categorical_columns),
],
remainder="passthrough",
)
return make_pipeline(preprocessor, outlier_detector)
以下 fit_predict 函式會傳回 X 的平均離群值分數。
from time import perf_counter
def fit_predict(estimator, X):
tic = perf_counter()
if estimator[-1].__class__.__name__ == "LocalOutlierFactor":
estimator.fit(X)
y_pred = estimator[-1].negative_outlier_factor_
else: # "IsolationForest"
y_pred = estimator.fit(X).decision_function(X)
toc = perf_counter()
print(f"Duration for {model_name}: {toc - tic:.2f} s")
return y_pred
在範例的其餘部分,我們每個區段處理一個資料集。在載入資料後,目標會被修改為由兩個類別組成:0 代表內圍值,1 代表離群值。由於 scikit-learn 文件計算的限制,某些資料集的樣本大小會使用分層 train_test_split 來縮減。
此外,我們設定 n_neighbors 以符合預期的異常數量 expected_n_anomalies = n_samples * expected_anomaly_fraction。這是一個很好的啟發式方法,只要離群值的比例不是非常低,原因在於 n_neighbors 應至少大於人口較少叢集中的樣本數 (請參閱 使用局部離群值因子 (LOF) 進行離群值偵測)。
KDDCup99 - SA 資料集#
Kddcup 99 資料集 是使用封閉網路和手動注入的攻擊產生的。SA 資料集是它的子集,其僅通過選擇所有正常資料和約 3% 的異常比例而獲得。
import numpy as np
from sklearn.datasets import fetch_kddcup99
from sklearn.model_selection import train_test_split
X, y = fetch_kddcup99(
subset="SA", percent10=True, random_state=42, return_X_y=True, as_frame=True
)
y = (y != b"normal.").astype(np.int32)
X, _, y, _ = train_test_split(X, y, train_size=0.1, stratify=y, random_state=42)
n_samples, anomaly_frac = X.shape[0], y.mean()
print(f"{n_samples} datapoints with {y.sum()} anomalies ({anomaly_frac:.02%})")
10065 datapoints with 338 anomalies (3.36%)
SA 資料集包含 41 個特徵,其中 3 個是類別特徵:「protocol_type」、「service」和「flag」。
y_true = {}
y_pred = {"LOF": {}, "IForest": {}}
model_names = ["LOF", "IForest"]
cat_columns = ["protocol_type", "service", "flag"]
y_true["KDDCup99 - SA"] = y
for model_name in model_names:
model = make_estimator(
name=model_name,
categorical_columns=cat_columns,
lof_kw={"n_neighbors": int(n_samples * anomaly_frac)},
iforest_kw={"random_state": 42},
)
y_pred[model_name]["KDDCup99 - SA"] = fit_predict(model, X)
Duration for LOF: 2.14 s
Duration for IForest: 0.31 s
森林覆蓋類型資料集#
森林覆蓋類型是一個多類別資料集,其中目標是給定一片森林中主要樹種。它包含 54 個特徵,其中一些特徵 (「Wilderness_Area」和「Soil_Type」) 已經以二進制編碼。雖然最初是作為分類任務,但可以將內圍值視為使用標籤 2 編碼的樣本,將離群值視為使用標籤 4 編碼的樣本。
from sklearn.datasets import fetch_covtype
X, y = fetch_covtype(return_X_y=True, as_frame=True)
s = (y == 2) + (y == 4)
X = X.loc[s]
y = y.loc[s]
y = (y != 2).astype(np.int32)
X, _, y, _ = train_test_split(X, y, train_size=0.05, stratify=y, random_state=42)
X_forestcover = X # save X for later use
n_samples, anomaly_frac = X.shape[0], y.mean()
print(f"{n_samples} datapoints with {y.sum()} anomalies ({anomaly_frac:.02%})")
14302 datapoints with 137 anomalies (0.96%)
y_true["forestcover"] = y
for model_name in model_names:
model = make_estimator(
name=model_name,
lof_kw={"n_neighbors": int(n_samples * anomaly_frac)},
iforest_kw={"random_state": 42},
)
y_pred[model_name]["forestcover"] = fit_predict(model, X)
Duration for LOF: 1.89 s
Duration for IForest: 0.26 s
埃姆斯房屋資料集#
埃姆斯房屋資料集最初是一個迴歸資料集,其中目標是愛荷華州埃姆斯房屋的銷售價格。在這裡,我們將每平方英尺價格超過 70 美元的房屋視為離群值,從而將其轉換為離群值偵測問題。為了使問題更容易,我們刪除了每平方英尺價格在 40 美元到 70 美元之間的中間價格。
import matplotlib.pyplot as plt
from sklearn.datasets import fetch_openml
X, y = fetch_openml(name="ames_housing", version=1, return_X_y=True, as_frame=True)
y = y.div(X["Lot_Area"])
# None values in pandas 1.5.1 were mapped to np.nan in pandas 2.0.1
X["Misc_Feature"] = X["Misc_Feature"].cat.add_categories("NoInfo").fillna("NoInfo")
X["Mas_Vnr_Type"] = X["Mas_Vnr_Type"].cat.add_categories("NoInfo").fillna("NoInfo")
X.drop(columns="Lot_Area", inplace=True)
mask = (y < 40) | (y > 70)
X = X.loc[mask]
y = y.loc[mask]
y.hist(bins=20, edgecolor="black")
plt.xlabel("House price in USD/sqft")
_ = plt.title("Distribution of house prices in Ames")
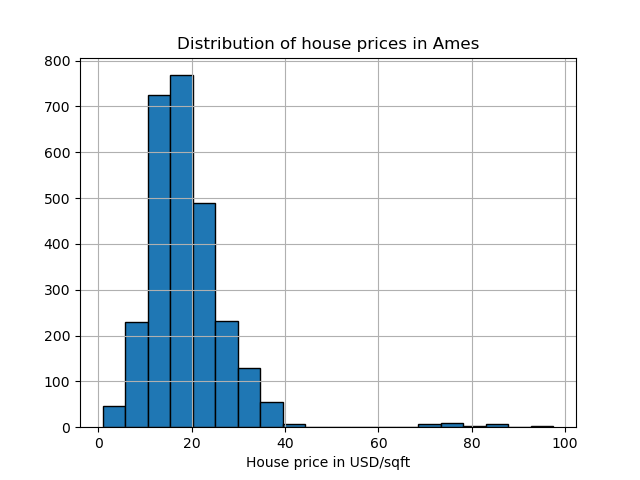
y = (y > 70).astype(np.int32)
n_samples, anomaly_frac = X.shape[0], y.mean()
print(f"{n_samples} datapoints with {y.sum()} anomalies ({anomaly_frac:.02%})")
2714 datapoints with 30 anomalies (1.11%)
該資料集包含 46 個類別特徵。在這種情況下,使用 make_column_selector 來尋找它們比傳遞手動製作的列表更容易。
from sklearn.compose import make_column_selector as selector
categorical_columns_selector = selector(dtype_include="category")
cat_columns = categorical_columns_selector(X)
y_true["ames_housing"] = y
for model_name in model_names:
model = make_estimator(
name=model_name,
categorical_columns=cat_columns,
lof_kw={"n_neighbors": int(n_samples * anomaly_frac)},
iforest_kw={"random_state": 42},
)
y_pred[model_name]["ames_housing"] = fit_predict(model, X)
Duration for LOF: 0.93 s
Duration for IForest: 0.24 s
心臟胎兒監護資料集#
「胎兒心電圖數據集」是一個多類別數據集,其中包含胎兒心電圖 (cardiotocograms),類別以 1 到 10 的標籤編碼,代表胎兒心率 (FHR) 模式。在此,我們將第 3 類(少數類別)設定為代表離群值。它包含 30 個數值特徵,其中一些是二元編碼,另一些是連續的。
X, y = fetch_openml(name="cardiotocography", version=1, return_X_y=True, as_frame=False)
X_cardiotocography = X # save X for later use
s = y == "3"
y = s.astype(np.int32)
n_samples, anomaly_frac = X.shape[0], y.mean()
print(f"{n_samples} datapoints with {y.sum()} anomalies ({anomaly_frac:.02%})")
2126 datapoints with 53 anomalies (2.49%)
y_true["cardiotocography"] = y
for model_name in model_names:
model = make_estimator(
name=model_name,
lof_kw={"n_neighbors": int(n_samples * anomaly_frac)},
iforest_kw={"random_state": 42},
)
y_pred[model_name]["cardiotocography"] = fit_predict(model, X)
Duration for LOF: 0.06 s
Duration for IForest: 0.16 s
繪製並解釋結果#
演算法的效能與偽陽性率 (FPR) 低時,真陽性率 (TPR) 有多好有關。最佳演算法在圖表的左上方有曲線,並且曲線下面積 (AUC) 接近 1。對角虛線代表離群值和正常值的隨機分類。
import math
from sklearn.metrics import RocCurveDisplay
cols = 2
pos_label = 0 # mean 0 belongs to positive class
datasets_names = y_true.keys()
rows = math.ceil(len(datasets_names) / cols)
fig, axs = plt.subplots(nrows=rows, ncols=cols, squeeze=False, figsize=(10, rows * 4))
for ax, dataset_name in zip(axs.ravel(), datasets_names):
for model_idx, model_name in enumerate(model_names):
display = RocCurveDisplay.from_predictions(
y_true[dataset_name],
y_pred[model_name][dataset_name],
pos_label=pos_label,
name=model_name,
ax=ax,
plot_chance_level=(model_idx == len(model_names) - 1),
chance_level_kw={"linestyle": ":"},
)
ax.set_title(dataset_name)
_ = plt.tight_layout(pad=2.0) # spacing between subplots
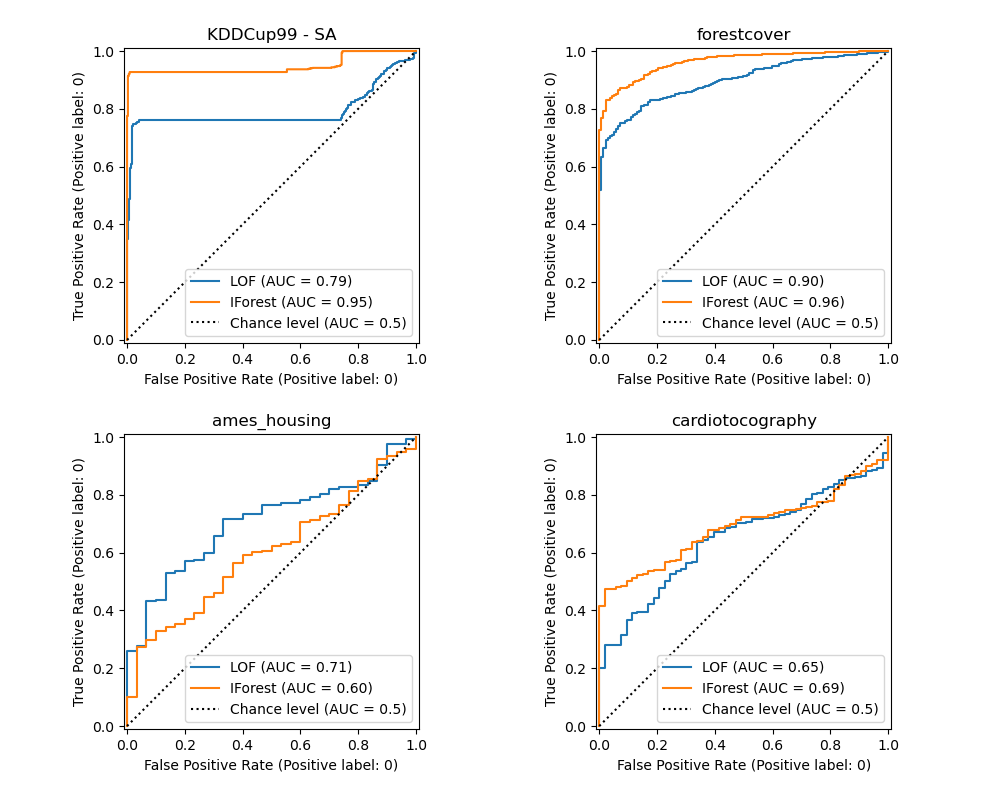
我們觀察到,一旦調整了鄰居數量,對於森林覆蓋和胎兒心電圖數據集,LOF 和 IForest 在 ROC AUC 方面表現相似。對於 SA 數據集,IForest 的分數略好,而 LOF 在 Ames 房屋數據集上的表現明顯優於 IForest。
然而,請回想一下,在具有大量樣本的數據集上,Isolation Forest 的訓練速度往往比 LOF 快得多。LOF 需要計算成對距離以找到最近鄰居,其複雜度與觀察值的數量呈二次方關係。這可能使此方法在大數據集上難以使用。
消融研究#
在本節中,我們探討超參數 n_neighbors 以及縮放 LOF 模型數值變數的選擇所造成的影響。在這裡,我們使用「森林覆蓋類型」數據集,因為二元編碼的類別引入了 0 到 1 之間歐幾里得距離的自然尺度。然後,我們希望使用一種縮放方法來避免給予非二元特徵特權,並且該方法對於離群值足夠穩健,以至於尋找它們的任務不會變得太困難。
X = X_forestcover
y = y_true["forestcover"]
n_samples = X.shape[0]
n_neighbors_list = (n_samples * np.array([0.2, 0.02, 0.01, 0.001])).astype(np.int32)
model = make_pipeline(RobustScaler(), LocalOutlierFactor())
linestyles = ["solid", "dashed", "dashdot", ":", (5, (10, 3))]
fig, ax = plt.subplots()
for model_idx, (linestyle, n_neighbors) in enumerate(zip(linestyles, n_neighbors_list)):
model.set_params(localoutlierfactor__n_neighbors=n_neighbors)
model.fit(X)
y_pred = model[-1].negative_outlier_factor_
display = RocCurveDisplay.from_predictions(
y,
y_pred,
pos_label=pos_label,
name=f"n_neighbors = {n_neighbors}",
ax=ax,
plot_chance_level=(model_idx == len(n_neighbors_list) - 1),
chance_level_kw={"linestyle": (0, (1, 10))},
linestyle=linestyle,
linewidth=2,
)
_ = ax.set_title("RobustScaler with varying n_neighbors\non forestcover dataset")
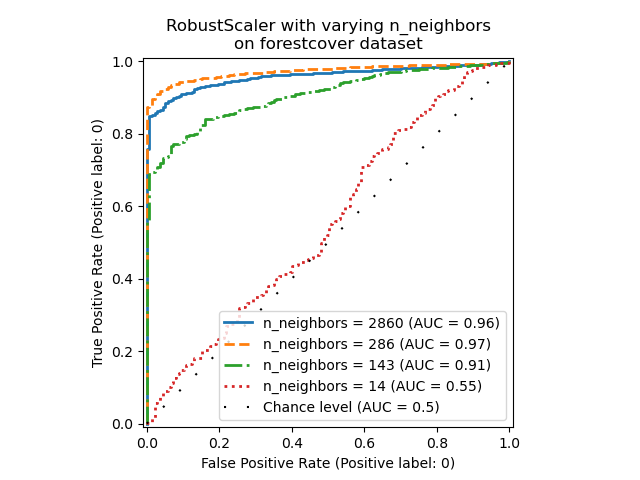
我們觀察到,鄰居數量對模型的效能有很大影響。如果可以存取(至少一些)真實標籤,則務必相應地調整 n_neighbors。一種方便的做法是探索 n_neighbors 的值,其數量級與預期的汙染程度相同。
from sklearn.preprocessing import MinMaxScaler, SplineTransformer, StandardScaler
preprocessor_list = [
None,
RobustScaler(),
StandardScaler(),
MinMaxScaler(),
SplineTransformer(),
]
expected_anomaly_fraction = 0.02
lof = LocalOutlierFactor(n_neighbors=int(n_samples * expected_anomaly_fraction))
fig, ax = plt.subplots()
for model_idx, (linestyle, preprocessor) in enumerate(
zip(linestyles, preprocessor_list)
):
model = make_pipeline(preprocessor, lof)
model.fit(X)
y_pred = model[-1].negative_outlier_factor_
display = RocCurveDisplay.from_predictions(
y,
y_pred,
pos_label=pos_label,
name=str(preprocessor).split("(")[0],
ax=ax,
plot_chance_level=(model_idx == len(preprocessor_list) - 1),
chance_level_kw={"linestyle": (0, (1, 10))},
linestyle=linestyle,
linewidth=2,
)
_ = ax.set_title("Fixed n_neighbors with varying preprocessing\non forestcover dataset")
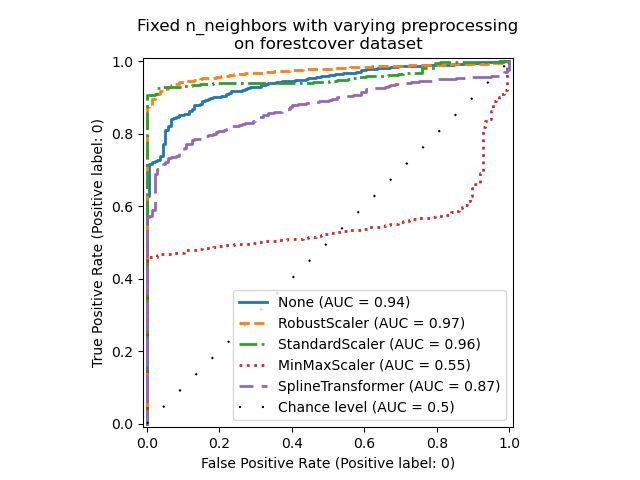
一方面,「RobustScaler」預設使用四分位數間距 (IQR) 來獨立縮放每個特徵,即資料的第 25 個和第 75 個百分位數之間的範圍。它通過減去中位數來中心化資料,然後通過除以 IQR 來縮放資料。IQR 對離群值具有穩健性:與範圍、平均值和標準差相比,中位數和四分位數間距受極端值的影響較小。此外,「RobustScaler」不像「StandardScaler」那樣擠壓邊緣離群值。
另一方面,「MinMaxScaler」會個別縮放每個特徵,使其範圍對應到零和一之間的範圍。如果資料中存在離群值,它們可能會將資料向最小值或最大值傾斜,導致具有較大邊緣離群值的資料分佈完全不同:所有非離群值可能因此幾乎被壓縮在一起。
我們還評估了完全不進行預處理(通過將 None 傳遞到管線中),「StandardScaler」和「SplineTransformer」。請參閱它們各自的文件以了解更多詳細資訊。
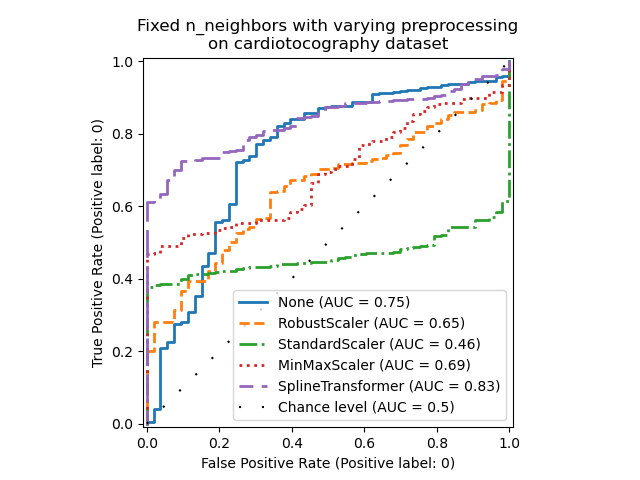
請注意,如下所示,最佳預處理取決於數據集
X = X_cardiotocography
y = y_true["cardiotocography"]
n_samples, expected_anomaly_fraction = X.shape[0], 0.025
lof = LocalOutlierFactor(n_neighbors=int(n_samples * expected_anomaly_fraction))
fig, ax = plt.subplots()
for model_idx, (linestyle, preprocessor) in enumerate(
zip(linestyles, preprocessor_list)
):
model = make_pipeline(preprocessor, lof)
model.fit(X)
y_pred = model[-1].negative_outlier_factor_
display = RocCurveDisplay.from_predictions(
y,
y_pred,
pos_label=pos_label,
name=str(preprocessor).split("(")[0],
ax=ax,
plot_chance_level=(model_idx == len(preprocessor_list) - 1),
chance_level_kw={"linestyle": (0, (1, 10))},
linestyle=linestyle,
linewidth=2,
)
ax.set_title(
"Fixed n_neighbors with varying preprocessing\non cardiotocography dataset"
)
plt.show()
腳本的總執行時間: (0 分鐘 52.650 秒)
相關範例