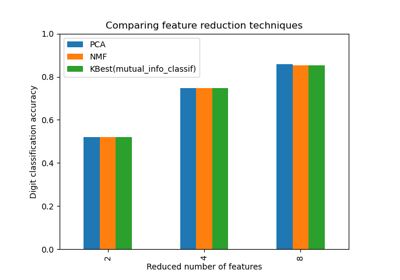
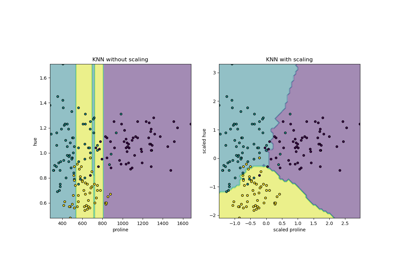
PCA#
- class sklearn.decomposition.PCA(n_components=None, *, copy=True, whiten=False, svd_solver='auto', tol=0.0, iterated_power='auto', n_oversamples=10, power_iteration_normalizer='auto', random_state=None)[原始碼]#
主成分分析 (PCA)。
使用資料的奇異值分解 (Singular Value Decomposition, SVD) 來進行線性降維,將資料投影到較低維度的空間。輸入資料在應用 SVD 之前會針對每個特徵進行中心化,但不會進行縮放。
它使用 LAPACK 實現的完整 SVD 或 Halko 等人在 2009 年提出的隨機截斷 SVD,具體取決於輸入資料的形狀和要提取的成分數量。
對於稀疏輸入,可以使用截斷 SVD 的 ARPACK 實現(即透過
scipy.sparse.linalg.svds)。或者,可以考慮使用TruncatedSVD,其中資料不會被中心化。請注意,此類別僅針對某些求解器(例如“arpack”和“covariance_eigh”)支援稀疏輸入。如需使用稀疏資料的替代方案,請參閱
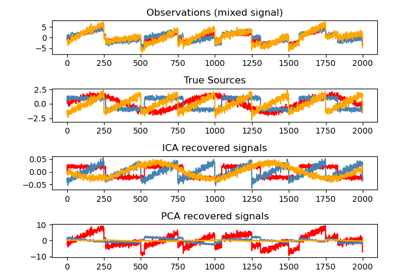
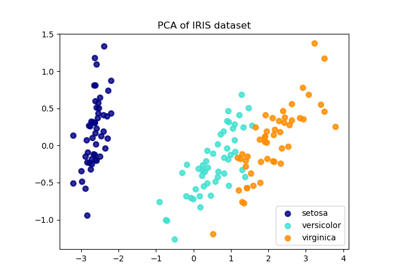
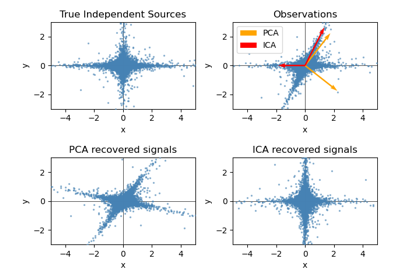
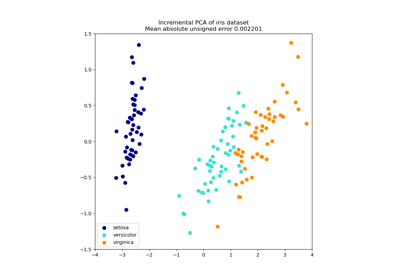
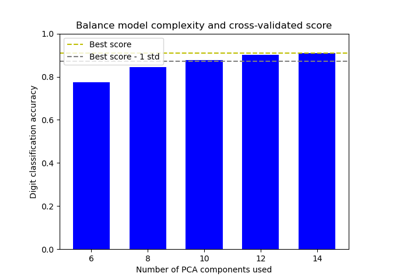
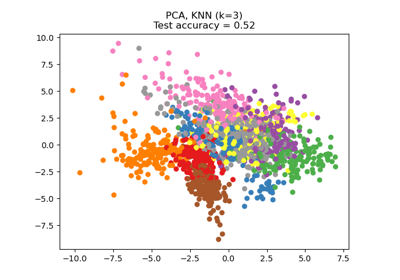
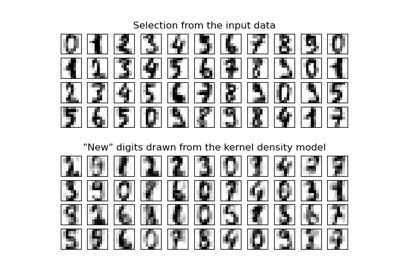
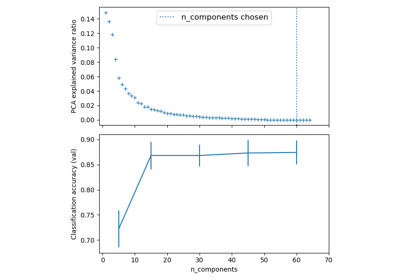
TruncatedSVD。如需使用範例,請參閱 在鳶尾花資料集上進行主成分分析 (PCA)
請在使用者指南中閱讀更多內容。
- 參數:
- n_componentsint、float 或 ‘mle’,預設值為 None
要保留的成分數量。如果未設定 n_components,則保留所有成分
n_components == min(n_samples, n_features)
如果
n_components == 'mle'且svd_solver == 'full',則使用 Minka 的 MLE 來猜測維度。使用n_components == 'mle'會將svd_solver == 'auto'解釋為svd_solver == 'full'。如果
0 < n_components < 1且svd_solver == 'full',則選擇成分的數量,以便需要解釋的變異量大於 n_components 指定的百分比。如果
svd_solver == 'arpack',則成分的數量必須嚴格小於 n_features 和 n_samples 的最小值。因此,None 的情況會導致
n_components == min(n_samples, n_features) - 1
- copybool,預設值為 True
如果為 False,則傳遞給 fit 的資料會被覆寫,並且執行 fit(X).transform(X) 不會產生預期的結果,請改用 fit_transform(X)。
- whitenbool,預設值為 False
當為 True(預設為 False)時,
components_向量會乘以 n_samples 的平方根,然後除以奇異值,以確保不相關的輸出具有單位成分式的變異數。白化會從轉換後的訊號中移除一些資訊(成分的相對變異數縮放),但有時可以透過使資料符合某些硬編碼的假設,來提高下游估計器的預測準確性。
- svd_solver{‘auto’, ‘full’, ‘covariance_eigh’, ‘arpack’, ‘randomized’},預設值為 ‘auto’
- “auto”
求解器由預設的 'auto' 原則選取,該原則基於
X.shape和n_components:如果輸入資料的特徵少於 1000 個,且樣本數是特徵數的 10 倍以上,則使用“covariance_eigh”求解器。否則,如果輸入資料大於 500x500,且要提取的成分數量低於資料最小維度的 80%,則選擇效率更高的“randomized”方法。否則,將計算精確的“full” SVD,並在之後選擇性地截斷。- “full”
執行精確的完整 SVD,透過
scipy.linalg.svd呼叫標準 LAPACK 求解器,並透過後處理選擇成分- “covariance_eigh”
預先計算共變異數矩陣(在中心化資料上),在共變異數矩陣上執行經典的特徵值分解,通常使用 LAPACK,並透過後處理選擇成分。此求解器對於 n_samples >> n_features 和小 n_features 非常有效。但是,對於大的 n_features(需要具體化共變異數矩陣的大記憶體空間),否則它是無法處理的。另請注意,與“full”求解器相比,此求解器有效地使條件數加倍,因此數值穩定性較差(例如,對於具有大範圍奇異值的輸入資料)。
- “arpack”
執行截斷到
n_components的 SVD,透過scipy.sparse.linalg.svds呼叫 ARPACK 求解器。它嚴格要求0 < n_components < min(X.shape)- “randomized”
透過 Halko 等人的方法執行隨機 SVD。
在 0.18.0 版本中新增。
在 1.5 版本中變更:新增了 ‘covariance_eigh’ 求解器。
- tolfloat,預設值為 0.0
svd_solver == ‘arpack’ 計算的奇異值的容差。必須在範圍 [0.0, infinity) 中。
在 0.18.0 版本中新增。
- iterated_powerint 或 ‘auto’,預設值為 ‘auto’
svd_solver == ‘randomized’ 計算的冪方法的迭代次數。必須在範圍 [0, infinity) 中。
在 0.18.0 版本中新增。
- n_oversamplesint,預設值為 10
此參數僅在
svd_solver="randomized"時相關。它對應於對X的範圍進行採樣以確保正確條件的其他隨機向量數量。如需更多詳細資訊,請參閱randomized_svd。在 1.1 版本中新增。
- power_iteration_normalizer{‘auto’, ‘QR’, ‘LU’, ‘none’}, 預設值=’auto’
隨機 SVD 解算器的冪迭代正規化器。ARPACK 不使用。詳情請參閱
randomized_svd。在 1.1 版本中新增。
- random_stateint、RandomState 實例或 None,預設值=None
當使用 ‘arpack’ 或 ‘randomized’ 解算器時使用。傳遞一個 int 以在多個函數呼叫中產生可重複的結果。請參閱詞彙表。
在 0.18.0 版本中新增。
- 屬性:
- components_形狀為 (n_components, n_features) 的 ndarray
特徵空間中的主軸,表示資料中最大變異的方向。等效地,中心輸入資料的右奇異向量,與其特徵向量平行。這些成分按照遞減的
explained_variance_排序。- explained_variance_形狀為 (n_components,) 的 ndarray
每個選定成分解釋的變異量。變異數估計使用
n_samples - 1自由度。等於 X 的共變異數矩陣的 n_components 個最大特徵值。
在 0.18 版中新增。
- explained_variance_ratio_形狀為 (n_components,) 的 ndarray
每個選定成分解釋的變異數百分比。
如果未設定
n_components,則會儲存所有成分,且比率總和等於 1.0。- singular_values_形狀為 (n_components,) 的 ndarray
對應於每個選定成分的奇異值。奇異值等於低維空間中
n_components變數的 2-範數。在 0.19 版中新增。
- mean_形狀為 (n_features,) 的 ndarray
從訓練集估計的每個特徵的經驗平均值。
等於
X.mean(axis=0)。- n_components_int
估計的成分數量。當 n_components 設定為 ‘mle’ 或介於 0 和 1 之間的數字(svd_solver == ‘full’ 時),此數字會從輸入資料估計。否則,它等於參數 n_components,或 n_features 和 n_samples 的較小值(如果 n_components 為 None)。
- n_samples_int
訓練資料中的樣本數。
- noise_variance_float
根據 Tipping 和 Bishop 1999 年的機率 PCA 模型估計的雜訊共變異數。請參閱 C. Bishop 的「Pattern Recognition and Machine Learning」,第 12.2.1 頁,第 574 頁,或 http://www.miketipping.com/papers/met-mppca.pdf。需要計算估計的資料共變異數和評分樣本。
等於 X 的共變異數矩陣的 (min(n_features, n_samples) - n_components) 個最小特徵值的平均值。
- n_features_in_int
在 fit 期間看到的特徵數量。
在 0.24 版中新增。
- feature_names_in_形狀為 (
n_features_in_,) 的 ndarray 在 fit 期間看到的特徵名稱。僅當
X具有全部為字串的特徵名稱時才定義。在 1.0 版中新增。
另請參閱
KernelPCAKernel 主成分分析。
SparsePCA稀疏主成分分析。
TruncatedSVD使用截斷 SVD 進行降維。
IncrementalPCA增量主成分分析。
參考文獻
對於 n_components == ‘mle’,此類別使用的方法來自:Minka, T. P.. “Automatic choice of dimensionality for PCA”. In NIPS, pp. 598-604
實作機率 PCA 模型,來自:Tipping, M. E., and Bishop, C. M. (1999). “Probabilistic principal component analysis”. Journal of the Royal Statistical Society: Series B (Statistical Methodology), 61(3), 611-622. 透過 score 和 score_samples 方法。
對於 svd_solver == ‘arpack’,請參閱
scipy.sparse.linalg.svds。對於 svd_solver == ‘randomized’,請參閱:Halko, N., Martinsson, P. G., and Tropp, J. A. (2011). “Finding structure with randomness: Probabilistic algorithms for constructing approximate matrix decompositions”. SIAM review, 53(2), 217-288. 以及 Martinsson, P. G., Rokhlin, V., and Tygert, M. (2011). “A randomized algorithm for the decomposition of matrices”. Applied and Computational Harmonic Analysis, 30(1), 47-68.
範例
>>> import numpy as np >>> from sklearn.decomposition import PCA >>> X = np.array([[-1, -1], [-2, -1], [-3, -2], [1, 1], [2, 1], [3, 2]]) >>> pca = PCA(n_components=2) >>> pca.fit(X) PCA(n_components=2) >>> print(pca.explained_variance_ratio_) [0.9924... 0.0075...] >>> print(pca.singular_values_) [6.30061... 0.54980...]
>>> pca = PCA(n_components=2, svd_solver='full') >>> pca.fit(X) PCA(n_components=2, svd_solver='full') >>> print(pca.explained_variance_ratio_) [0.9924... 0.00755...] >>> print(pca.singular_values_) [6.30061... 0.54980...]
>>> pca = PCA(n_components=1, svd_solver='arpack') >>> pca.fit(X) PCA(n_components=1, svd_solver='arpack') >>> print(pca.explained_variance_ratio_) [0.99244...] >>> print(pca.singular_values_) [6.30061...]
- fit(X, y=None)[原始碼]#
使用 X 來擬合模型。
- 參數:
- X形狀為 (n_samples, n_features) 的 {類陣列, 稀疏矩陣}
訓練資料,其中
n_samples是樣本數,而n_features是特徵數。- y忽略
忽略。
- 傳回:
- self物件
傳回實例本身。
- fit_transform(X, y=None)[原始碼]#
使用 X 擬合模型,並對 X 應用降維。
- 參數:
- X形狀為 (n_samples, n_features) 的 {類陣列, 稀疏矩陣}
訓練資料,其中
n_samples是樣本數,而n_features是特徵數。- y忽略
忽略。
- 傳回:
- X_new形狀為 (n_samples, n_components) 的 ndarray
轉換後的值。
附註
此方法會傳回 Fortran 排序的陣列。若要將其轉換為 C 排序的陣列,請使用 ‘np.ascontiguousarray’。
- get_covariance()[原始碼]#
使用產生模型計算資料共變異數。
cov = components_.T * S**2 * components_ + sigma2 * eye(n_features),其中 S**2 包含解釋的變異數,而 sigma2 包含雜訊變異數。- 傳回:
- cov形狀為 (n_features, n_features) 的陣列
資料的估計共變異數。
- get_feature_names_out(input_features=None)[原始碼]#
取得轉換的輸出特徵名稱。
輸出的特徵名稱會以小寫的類別名稱作為前綴。例如,如果轉換器輸出 3 個特徵,則輸出的特徵名稱為:
["class_name0", "class_name1", "class_name2"]。- 參數:
- input_features字串或 None 的類陣列,預設值=None
僅用於驗證特徵名稱是否與
fit中看到的名稱相同。
- 傳回:
- feature_names_out字串物件的 ndarray
轉換後的特徵名稱。
- get_metadata_routing()[原始碼]#
取得此物件的中繼資料路由。
請查看 使用者指南,了解路由機制如何運作。
- 傳回:
- routingMetadataRequest
封裝路由資訊的
MetadataRequest。
- get_params(deep=True)[原始碼]#
取得此估算器的參數。
- 參數:
- deep布林值,預設值=True
如果為 True,則會傳回此估算器和包含的子物件 (屬於估算器) 的參數。
- 傳回:
- params字典
參數名稱對應到它們的值。
- get_precision()[原始碼]#
使用產生模型計算資料精確度矩陣。
等於共變異數的反矩陣,但使用矩陣反轉引理計算以提高效率。
- 傳回:
- precision形狀為 (n_features, n_features) 的陣列
資料的估計精確度。
- inverse_transform(X)[原始碼]#
將資料轉換回其原始空間。
換句話說,傳回其轉換會是 X 的輸入
X_original。- 參數:
- X形狀為 (n_samples, n_components) 的類陣列
新資料,其中
n_samples是樣本數,而n_components是成分數。
- 傳回:
- X_original 形狀為 (n_samples, n_features) 的類陣列
原始資料,其中
n_samples是樣本數,而n_features是特徵數。
附註
如果啟用白化,inverse_transform 會計算確切的反向運算,其中包含反轉白化。
- score(X, y=None)[原始碼]#
傳回所有樣本的平均對數概似。
參見 C. Bishop 的《Pattern Recognition and Machine Learning》12.2.1 節第 574 頁,或 http://www.miketipping.com/papers/met-mppca.pdf
- 參數:
- X形狀為 (n_samples, n_features) 的類陣列 (array-like)
資料。
- y忽略
忽略。
- 傳回:
- ll浮點數 (float)
目前模型下樣本的平均對數似然率。
- score_samples(X)[來源]#
返回每個樣本的對數似然率。
參見 C. Bishop 的《Pattern Recognition and Machine Learning》12.2.1 節第 574 頁,或 http://www.miketipping.com/papers/met-mppca.pdf
- 參數:
- X形狀為 (n_samples, n_features) 的類陣列 (array-like)
資料。
- 傳回:
- ll形狀為 (n_samples,) 的 ndarray
目前模型下每個樣本的對數似然率。
- set_output(*, transform=None)[來源]#
設定輸出容器。
請參閱 介紹 set_output API 以了解如何使用此 API 的範例。
- 參數:
- transform{"default", "pandas", "polars"}, 預設值=None
配置
transform和fit_transform的輸出。"default":轉換器的預設輸出格式"pandas":DataFrame 輸出"polars":Polars 輸出None:轉換配置保持不變
1.4 版本新增:新增
"polars"選項。
- 傳回:
- self估算器實例
估算器實例。