注意
前往結尾以下載完整的範例程式碼。或透過 JupyterLite 或 Binder 在您的瀏覽器中執行此範例
特徵縮放的重要性#
透過標準化進行特徵縮放,也稱為 Z 分數正規化,是許多機器學習演算法的重要預處理步驟。它涉及重新縮放每個特徵,使其標準差為 1,平均值為 0。
即使基於樹的模型(幾乎)不受縮放的影響,許多其他演算法仍需要正規化特徵,通常出於不同的原因:為了簡化收斂(例如非懲罰邏輯回歸),以建立與未縮放資料的擬合完全不同的模型擬合(例如 KNeighbors 模型)。後者在本範例的第一部分中示範。
在本範例的第二部分中,我們展示了主成分分析 (PCA) 如何受到特徵正規化的影響。為了說明這一點,我們比較了使用PCA在未縮放資料上找到的主成分與首先使用StandardScaler縮放資料時獲得的主成分。
在本範例的最後一部分中,我們展示了正規化對在 PCA 縮減資料上訓練的模型準確性的影響。
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
載入並準備資料#
使用的資料集是 UCI 提供的葡萄酒識別資料集。此資料集具有連續特徵,這些特徵由於測量的不同屬性(例如酒精含量和蘋果酸)而在規模上是異質的。
from sklearn.datasets import load_wine
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
X, y = load_wine(return_X_y=True, as_frame=True)
scaler = StandardScaler().set_output(transform="pandas")
X_train, X_test, y_train, y_test = train_test_split(
X, y, test_size=0.30, random_state=42
)
scaled_X_train = scaler.fit_transform(X_train)
重新縮放對 k 近鄰模型的影響#
為了視覺化KNeighborsClassifier的決策邊界,在本節中,我們選擇一個具有不同數量級值的 2 個特徵的子集。
請記住,使用特徵的子集來訓練模型很可能會遺漏具有高預測影響的特徵,導致決策邊界比在完整特徵集上訓練的模型差得多。
import matplotlib.pyplot as plt
from sklearn.inspection import DecisionBoundaryDisplay
from sklearn.neighbors import KNeighborsClassifier
X_plot = X[["proline", "hue"]]
X_plot_scaled = scaler.fit_transform(X_plot)
clf = KNeighborsClassifier(n_neighbors=20)
def fit_and_plot_model(X_plot, y, clf, ax):
clf.fit(X_plot, y)
disp = DecisionBoundaryDisplay.from_estimator(
clf,
X_plot,
response_method="predict",
alpha=0.5,
ax=ax,
)
disp.ax_.scatter(X_plot["proline"], X_plot["hue"], c=y, s=20, edgecolor="k")
disp.ax_.set_xlim((X_plot["proline"].min(), X_plot["proline"].max()))
disp.ax_.set_ylim((X_plot["hue"].min(), X_plot["hue"].max()))
return disp.ax_
fig, (ax1, ax2) = plt.subplots(ncols=2, figsize=(12, 6))
fit_and_plot_model(X_plot, y, clf, ax1)
ax1.set_title("KNN without scaling")
fit_and_plot_model(X_plot_scaled, y, clf, ax2)
ax2.set_xlabel("scaled proline")
ax2.set_ylabel("scaled hue")
_ = ax2.set_title("KNN with scaling")
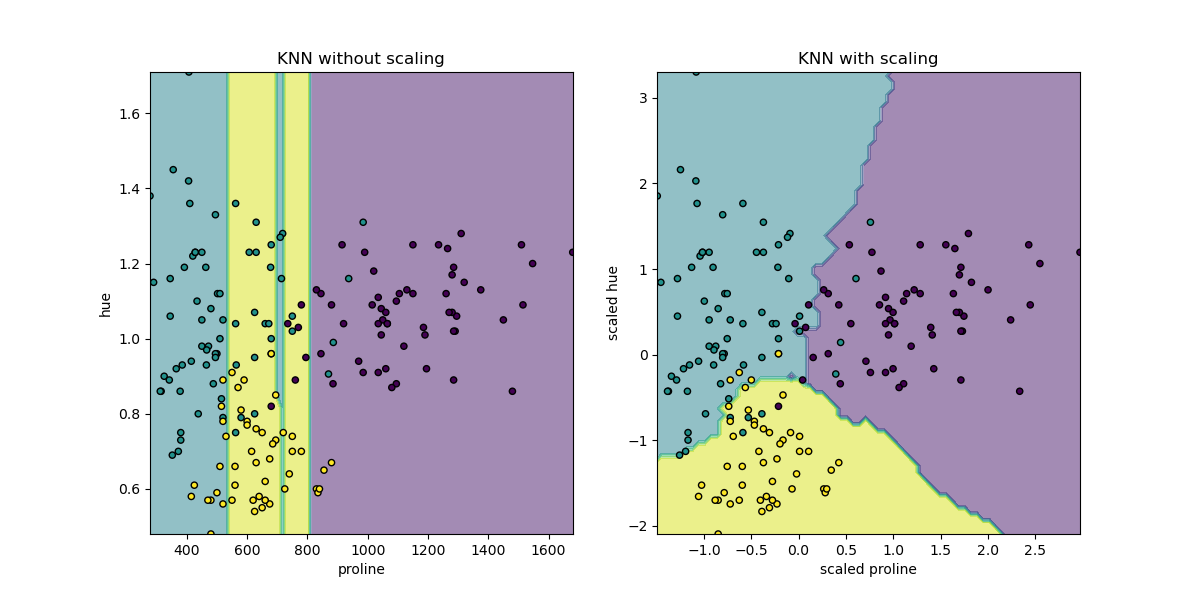
此處的決策邊界顯示,擬合縮放或未縮放的資料會導致完全不同的模型。原因是變數「proline」的值在 0 到 1,000 之間變化;而變數「hue」在 1 到 10 之間變化。因此,樣本之間的距離主要受到「proline」值差異的影響,而「hue」的值將被相對忽略。如果使用StandardScaler來正規化此資料庫,則兩個縮放值都大約在 -3 到 3 之間,並且鄰近結構將或多或少受到兩個變數的同等影響。
重新縮放對 PCA 降維的影響#
使用PCA進行降維包括找到最大化變異數的特徵。如果一個特徵僅僅由於其各自的縮放而比其他特徵變化更大,則PCA將確定該特徵主導主成分的方向。
我們可以檢查使用所有原始特徵的第一個主成分
import pandas as pd
from sklearn.decomposition import PCA
pca = PCA(n_components=2).fit(X_train)
scaled_pca = PCA(n_components=2).fit(scaled_X_train)
X_train_transformed = pca.transform(X_train)
X_train_std_transformed = scaled_pca.transform(scaled_X_train)
first_pca_component = pd.DataFrame(
pca.components_[0], index=X.columns, columns=["without scaling"]
)
first_pca_component["with scaling"] = scaled_pca.components_[0]
first_pca_component.plot.bar(
title="Weights of the first principal component", figsize=(6, 8)
)
_ = plt.tight_layout()
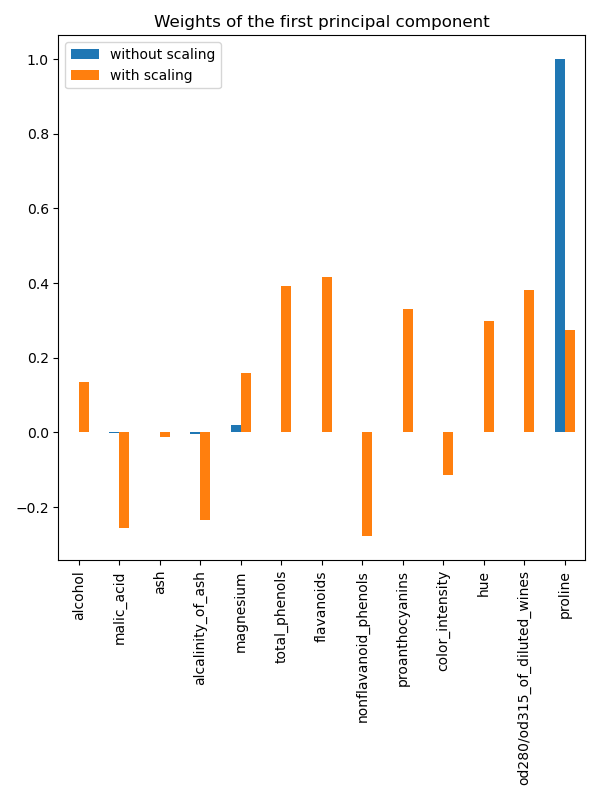
事實上,我們發現「proline」特徵在沒有縮放的情況下主導了第一個主成分的方向,比其他特徵高出約兩個數量級。當觀察縮放版本的資料的第一個主成分時,情況會有所不同,其中所有特徵的數量級大致相同。
我們可以視覺化兩種情況下主成分的分佈
fig, (ax1, ax2) = plt.subplots(nrows=1, ncols=2, figsize=(10, 5))
target_classes = range(0, 3)
colors = ("blue", "red", "green")
markers = ("^", "s", "o")
for target_class, color, marker in zip(target_classes, colors, markers):
ax1.scatter(
x=X_train_transformed[y_train == target_class, 0],
y=X_train_transformed[y_train == target_class, 1],
color=color,
label=f"class {target_class}",
alpha=0.5,
marker=marker,
)
ax2.scatter(
x=X_train_std_transformed[y_train == target_class, 0],
y=X_train_std_transformed[y_train == target_class, 1],
color=color,
label=f"class {target_class}",
alpha=0.5,
marker=marker,
)
ax1.set_title("Unscaled training dataset after PCA")
ax2.set_title("Standardized training dataset after PCA")
for ax in (ax1, ax2):
ax.set_xlabel("1st principal component")
ax.set_ylabel("2nd principal component")
ax.legend(loc="upper right")
ax.grid()
_ = plt.tight_layout()
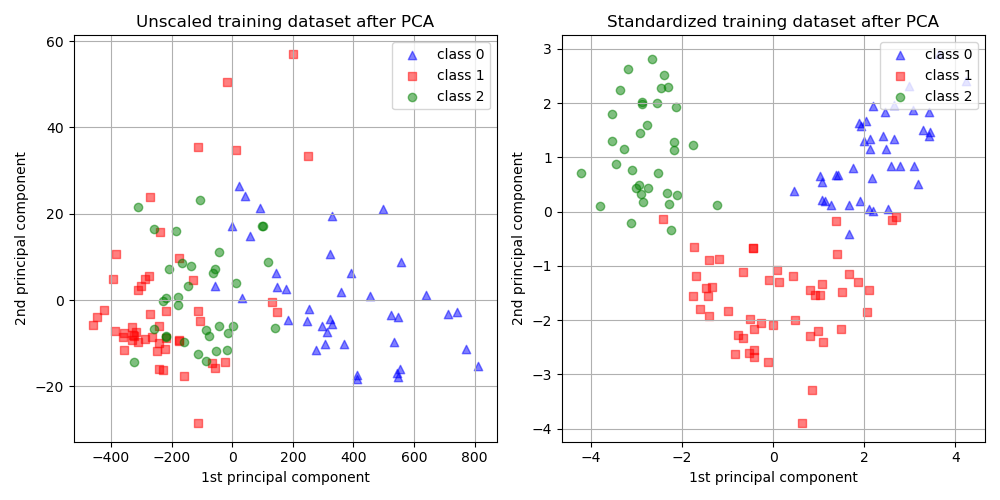
從上面的圖中,我們觀察到在降低維度之前縮放特徵會產生數量級相同的成分。在這種情況下,它還提高了類別的可分離性。事實上,在下一節中,我們確認更好的可分離性對整體模型效能有良好的影響。
重新縮放對模型效能的影響#
首先,我們展示了LogisticRegressionCV的最佳正規化如何取決於資料的縮放或非縮放
import numpy as np
from sklearn.linear_model import LogisticRegressionCV
from sklearn.pipeline import make_pipeline
Cs = np.logspace(-5, 5, 20)
unscaled_clf = make_pipeline(pca, LogisticRegressionCV(Cs=Cs))
unscaled_clf.fit(X_train, y_train)
scaled_clf = make_pipeline(scaler, pca, LogisticRegressionCV(Cs=Cs))
scaled_clf.fit(X_train, y_train)
print(f"Optimal C for the unscaled PCA: {unscaled_clf[-1].C_[0]:.4f}\n")
print(f"Optimal C for the standardized data with PCA: {scaled_clf[-1].C_[0]:.2f}")
Optimal C for the unscaled PCA: 0.0004
Optimal C for the standardized data with PCA: 20.69
對於在套用 PCA 之前未縮放的資料,正規化的需求較高(C的值較低)。我們現在評估縮放對最佳模型的準確性和平均對數損失的影響
from sklearn.metrics import accuracy_score, log_loss
y_pred = unscaled_clf.predict(X_test)
y_pred_scaled = scaled_clf.predict(X_test)
y_proba = unscaled_clf.predict_proba(X_test)
y_proba_scaled = scaled_clf.predict_proba(X_test)
print("Test accuracy for the unscaled PCA")
print(f"{accuracy_score(y_test, y_pred):.2%}\n")
print("Test accuracy for the standardized data with PCA")
print(f"{accuracy_score(y_test, y_pred_scaled):.2%}\n")
print("Log-loss for the unscaled PCA")
print(f"{log_loss(y_test, y_proba):.3}\n")
print("Log-loss for the standardized data with PCA")
print(f"{log_loss(y_test, y_proba_scaled):.3}")
Test accuracy for the unscaled PCA
35.19%
Test accuracy for the standardized data with PCA
96.30%
Log-loss for the unscaled PCA
0.957
Log-loss for the standardized data with PCA
0.0825
當資料在PCA之前縮放時,觀察到預測準確性存在明顯差異,因為它大大優於未縮放版本。這與從上一節的圖中獲得的直觀概念一致,其中當在使用PCA之前縮放時,組件會變成線性可分離的。
請注意,在這種情況下,具有縮放特徵的模型比具有未縮放特徵的模型表現更好,因為預期所有變數都具有預測性,而且我們寧願避免其中一些變數被相對忽略。
如果較小規模的變數不具有預測性,則在縮放特徵後可能會導致效能下降:雜訊特徵在縮放後將對預測做出更多貢獻,因此縮放將增加過度擬合。
最後但並非最不重要的一點是,我們觀察到透過縮放步驟可以獲得較低的對數損失。
腳本總執行時間: (0 分鐘 1.966 秒)
相關範例