注意
跳到結尾以下載完整的範例程式碼。或透過 JupyterLite 或 Binder 在您的瀏覽器中執行此範例
主成分迴歸與偏最小平方迴歸#
此範例比較玩具資料集上的主成分迴歸 (PCR) 和偏最小平方迴歸 (PLS)。我們的目標是說明當目標與資料中一些具有低變異數的方向高度相關時,PLS 如何優於 PCR。
PCR 是由兩個步驟組成的迴歸器:首先,將PCA套用至訓練資料,可能會執行降維;然後,在轉換後的樣本上訓練迴歸器 (例如線性迴歸器)。在PCA中,轉換是完全非監督的,這表示不會使用關於目標的資訊。因此,在某些目標與具有低變異數的方向高度相關的資料集中,PCR 的效能可能不佳。事實上,PCA 的降維將資料投影到較低維度的空間,其中沿著每個軸貪婪地最大化投影資料的變異數。儘管它們對目標具有最高的預測能力,但變異數較低的方向將會被捨棄,而最終的迴歸器將無法利用它們。
PLS 既是轉換器又是迴歸器,它與 PCR 非常相似:它也會在將線性迴歸器套用到轉換後的資料之前,對樣本執行降維。與 PCR 的主要區別在於 PLS 轉換是監督的。因此,正如我們將在此範例中看到的,它不會受到我們剛才提到的問題影響。
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
資料#
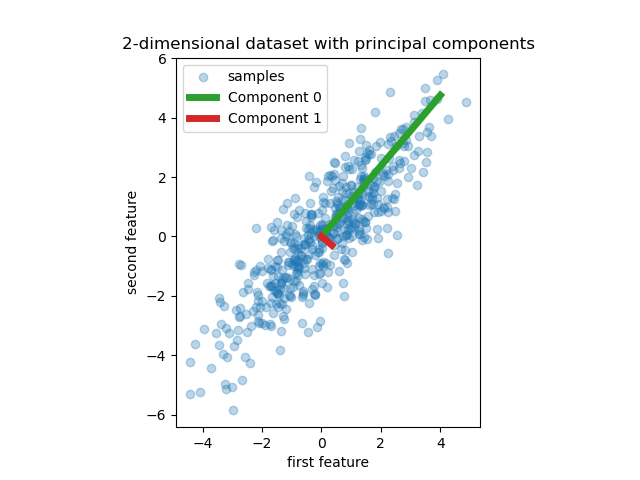
我們首先建立一個具有兩個特徵的簡單資料集。在我們深入研究 PCR 和 PLS 之前,我們先擬合 PCA 估計器以顯示此資料集的兩個主成分,也就是說,解釋資料中最多變異數的兩個方向。
import matplotlib.pyplot as plt
import numpy as np
from sklearn.decomposition import PCA
rng = np.random.RandomState(0)
n_samples = 500
cov = [[3, 3], [3, 4]]
X = rng.multivariate_normal(mean=[0, 0], cov=cov, size=n_samples)
pca = PCA(n_components=2).fit(X)
plt.scatter(X[:, 0], X[:, 1], alpha=0.3, label="samples")
for i, (comp, var) in enumerate(zip(pca.components_, pca.explained_variance_)):
comp = comp * var # scale component by its variance explanation power
plt.plot(
[0, comp[0]],
[0, comp[1]],
label=f"Component {i}",
linewidth=5,
color=f"C{i + 2}",
)
plt.gca().set(
aspect="equal",
title="2-dimensional dataset with principal components",
xlabel="first feature",
ylabel="second feature",
)
plt.legend()
plt.show()
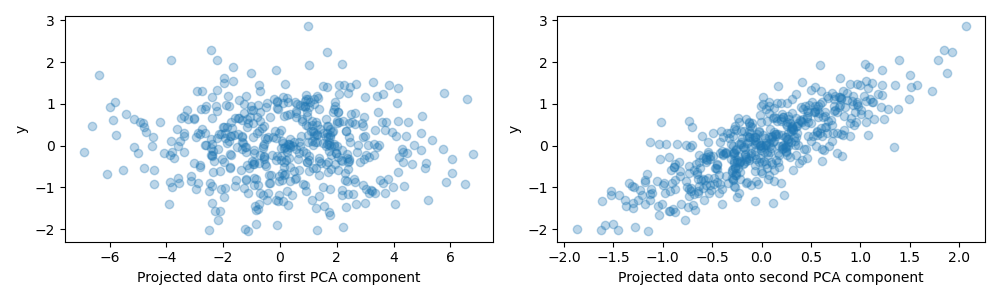
為了此範例的目的,我們現在定義目標 y,使其與具有小變異數的方向高度相關。為此,我們將 X 投影到第二個成分上,並在其中加入一些雜訊。
y = X.dot(pca.components_[1]) + rng.normal(size=n_samples) / 2
fig, axes = plt.subplots(1, 2, figsize=(10, 3))
axes[0].scatter(X.dot(pca.components_[0]), y, alpha=0.3)
axes[0].set(xlabel="Projected data onto first PCA component", ylabel="y")
axes[1].scatter(X.dot(pca.components_[1]), y, alpha=0.3)
axes[1].set(xlabel="Projected data onto second PCA component", ylabel="y")
plt.tight_layout()
plt.show()
在一個成分上的投影和預測能力#
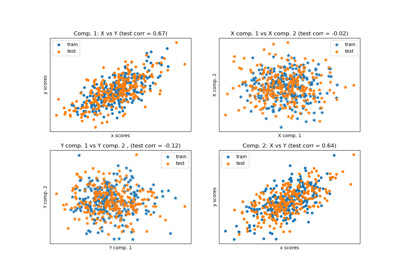
我們現在建立兩個迴歸器:PCR 和 PLS,並且為了我們的說明目的,我們將成分數設定為 1。在將資料饋送到 PCR 的 PCA 步驟之前,我們首先將其標準化,這符合良好實務建議。PLS 估計器具有內建的縮放功能。
對於這兩個模型,我們繪製投影到第一個成分上的資料與目標的關係。在這兩種情況下,此投影資料都是迴歸器將用作訓練資料的內容。
from sklearn.cross_decomposition import PLSRegression
from sklearn.decomposition import PCA
from sklearn.linear_model import LinearRegression
from sklearn.model_selection import train_test_split
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import StandardScaler
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=rng)
pcr = make_pipeline(StandardScaler(), PCA(n_components=1), LinearRegression())
pcr.fit(X_train, y_train)
pca = pcr.named_steps["pca"] # retrieve the PCA step of the pipeline
pls = PLSRegression(n_components=1)
pls.fit(X_train, y_train)
fig, axes = plt.subplots(1, 2, figsize=(10, 3))
axes[0].scatter(pca.transform(X_test), y_test, alpha=0.3, label="ground truth")
axes[0].scatter(
pca.transform(X_test), pcr.predict(X_test), alpha=0.3, label="predictions"
)
axes[0].set(
xlabel="Projected data onto first PCA component", ylabel="y", title="PCR / PCA"
)
axes[0].legend()
axes[1].scatter(pls.transform(X_test), y_test, alpha=0.3, label="ground truth")
axes[1].scatter(
pls.transform(X_test), pls.predict(X_test), alpha=0.3, label="predictions"
)
axes[1].set(xlabel="Projected data onto first PLS component", ylabel="y", title="PLS")
axes[1].legend()
plt.tight_layout()
plt.show()
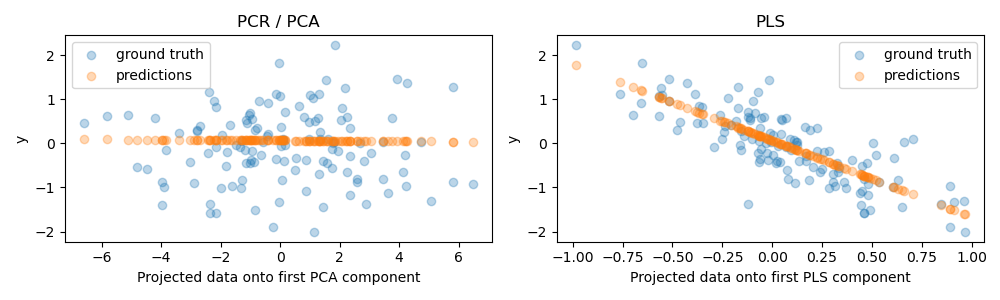
如預期的那樣,PCR 的非監督 PCA 轉換捨棄了第二個成分,也就是說,變異數最低的方向,儘管它是預測性最高的方向。這是因為 PCA 是一種完全非監督的轉換,並導致投影資料對目標的預測能力較低。
另一方面,PLS 迴歸器透過在轉換期間使用目標資訊,設法捕捉到變異數最低的方向的影響:它可以辨識出此方向實際上是預測性最高的。我們注意到,第一個 PLS 成分與目標呈負相關,這來自於特徵向量的符號是任意的。
我們也列印兩個估計器的 R 平方分數,這進一步證實了在這種情況下,PLS 是比 PCR 更好的替代方案。負 R 平方表示 PCR 的效能比僅預測目標平均值的迴歸器差。
print(f"PCR r-squared {pcr.score(X_test, y_test):.3f}")
print(f"PLS r-squared {pls.score(X_test, y_test):.3f}")
PCR r-squared -0.026
PLS r-squared 0.658
最後,我們注意到具有 2 個成分的 PCR 與 PLS 的效能相同:這是因為在這種情況下,PCR 能夠利用對目標具有最高預測能力的第二個成分。
pca_2 = make_pipeline(PCA(n_components=2), LinearRegression())
pca_2.fit(X_train, y_train)
print(f"PCR r-squared with 2 components {pca_2.score(X_test, y_test):.3f}")
PCR r-squared with 2 components 0.673
腳本的總執行時間: (0 分鐘 0.527 秒)
相關範例