注意
跳至結尾以下載完整的範例程式碼。或透過 JupyterLite 或 Binder 在您的瀏覽器中執行此範例
核函數 PCA#
此範例顯示主成分分析 (PCA) 與其核函數化版本 (KernelPCA) 之間的差異。
一方面,我們展示了 KernelPCA 能夠找到資料的投影,使其線性分離,而 PCA 則不能。
最後,我們展示了反轉此投影是使用 KernelPCA 的近似值,而使用 PCA 則是精確的。
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
投影資料:PCA 與 KernelPCA#
在本節中,我們展示了在使用主成分分析 (PCA) 投影資料時使用核函數的優點。我們建立一個由兩個巢狀圓組成的資料集。
from sklearn.datasets import make_circles
from sklearn.model_selection import train_test_split
X, y = make_circles(n_samples=1_000, factor=0.3, noise=0.05, random_state=0)
X_train, X_test, y_train, y_test = train_test_split(X, y, stratify=y, random_state=0)
讓我們快速看一下產生的資料集。
import matplotlib.pyplot as plt
_, (train_ax, test_ax) = plt.subplots(ncols=2, sharex=True, sharey=True, figsize=(8, 4))
train_ax.scatter(X_train[:, 0], X_train[:, 1], c=y_train)
train_ax.set_ylabel("Feature #1")
train_ax.set_xlabel("Feature #0")
train_ax.set_title("Training data")
test_ax.scatter(X_test[:, 0], X_test[:, 1], c=y_test)
test_ax.set_xlabel("Feature #0")
_ = test_ax.set_title("Testing data")
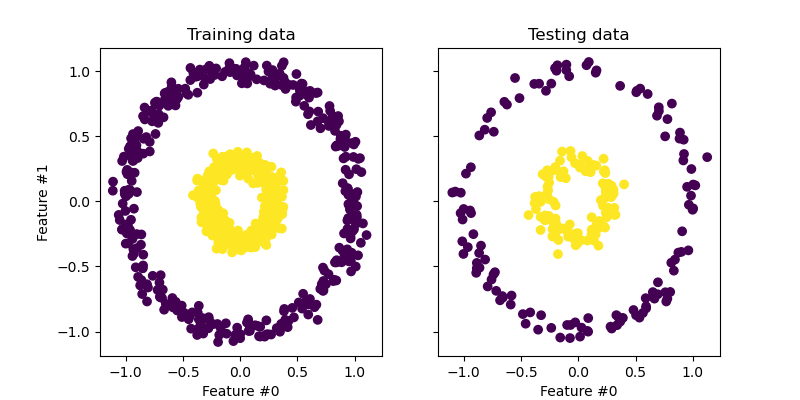
每個類別的樣本不能線性分離:沒有直線可以將內集樣本與外集樣本分開。
現在,我們將使用帶有和不帶有核函數的 PCA 來查看使用此類核函數的效果。此處使用的核函數是徑向基函數 (RBF) 核函數。
fig, (orig_data_ax, pca_proj_ax, kernel_pca_proj_ax) = plt.subplots(
ncols=3, figsize=(14, 4)
)
orig_data_ax.scatter(X_test[:, 0], X_test[:, 1], c=y_test)
orig_data_ax.set_ylabel("Feature #1")
orig_data_ax.set_xlabel("Feature #0")
orig_data_ax.set_title("Testing data")
pca_proj_ax.scatter(X_test_pca[:, 0], X_test_pca[:, 1], c=y_test)
pca_proj_ax.set_ylabel("Principal component #1")
pca_proj_ax.set_xlabel("Principal component #0")
pca_proj_ax.set_title("Projection of testing data\n using PCA")
kernel_pca_proj_ax.scatter(X_test_kernel_pca[:, 0], X_test_kernel_pca[:, 1], c=y_test)
kernel_pca_proj_ax.set_ylabel("Principal component #1")
kernel_pca_proj_ax.set_xlabel("Principal component #0")
_ = kernel_pca_proj_ax.set_title("Projection of testing data\n using KernelPCA")
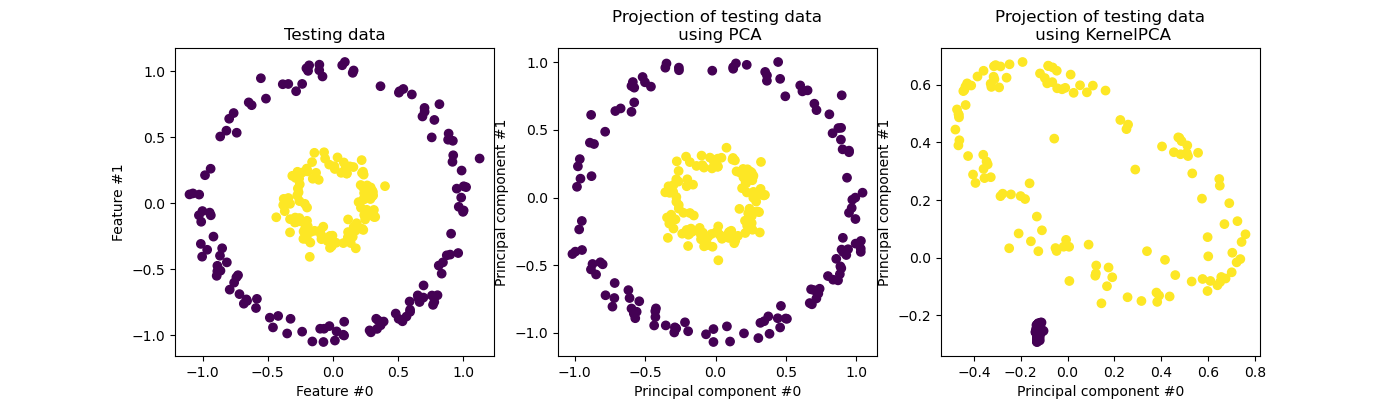
我們回顧一下,PCA 會線性轉換資料。直覺上,這表示座標系統將會置中、根據每個元件的變異數重新縮放,最後旋轉。此轉換獲得的資料是等向性的,現在可以投影到其主成分上。
因此,查看使用 PCA 進行的投影(即中間的圖),我們看到縮放沒有任何變化;實際上,資料是兩個以零為中心的同心圓,原始資料已經是等向性的。但是,我們可以觀察到資料已旋轉。總而言之,我們看到如果定義一個線性分類器來區分兩個類別的樣本,這樣的投影將無濟於事。
使用核函數可以進行非線性投影。在此,透過使用 RBF 核函數,我們期望投影將展開資料集,同時大約保留原始空間中彼此靠近的資料點對之間的相對距離。
我們在右側的圖中觀察到這種行為:給定類別的樣本彼此之間的距離比相反類別的樣本之間的距離更近,從而解開了兩個樣本集。現在,我們可以使用線性分類器來分離兩個類別的樣本。
投影到原始特徵空間#
使用 KernelPCA 時要記住的一個特殊性與重建(即原始特徵空間中的反向投影)有關。使用 PCA,如果 n_components 與原始特徵的數量相同,則重建將是精確的。此範例正是如此。
我們可以研究一下,當使用 KernelPCA 反向投影時是否會得到原始資料集。
X_reconstructed_pca = pca.inverse_transform(pca.transform(X_test))
X_reconstructed_kernel_pca = kernel_pca.inverse_transform(kernel_pca.transform(X_test))
fig, (orig_data_ax, pca_back_proj_ax, kernel_pca_back_proj_ax) = plt.subplots(
ncols=3, sharex=True, sharey=True, figsize=(13, 4)
)
orig_data_ax.scatter(X_test[:, 0], X_test[:, 1], c=y_test)
orig_data_ax.set_ylabel("Feature #1")
orig_data_ax.set_xlabel("Feature #0")
orig_data_ax.set_title("Original test data")
pca_back_proj_ax.scatter(X_reconstructed_pca[:, 0], X_reconstructed_pca[:, 1], c=y_test)
pca_back_proj_ax.set_xlabel("Feature #0")
pca_back_proj_ax.set_title("Reconstruction via PCA")
kernel_pca_back_proj_ax.scatter(
X_reconstructed_kernel_pca[:, 0], X_reconstructed_kernel_pca[:, 1], c=y_test
)
kernel_pca_back_proj_ax.set_xlabel("Feature #0")
_ = kernel_pca_back_proj_ax.set_title("Reconstruction via KernelPCA")
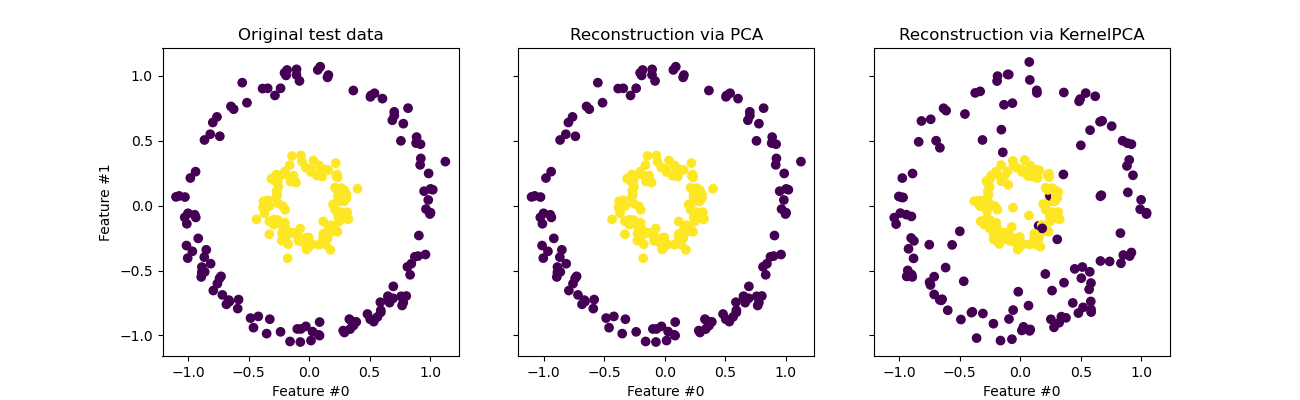
雖然我們看到 PCA 的完美重建,但我們觀察到 KernelPCA 的不同結果。
事實上,inverse_transform 無法依賴分析反向投影,因此無法進行精確重建。相反,會對 KernelRidge 進行內部訓練,以學習從核函數化 PCA 基礎到原始特徵空間的映射。因此,此方法會帶來近似值,在反向投影到原始特徵空間時會產生小的差異。
為了使用 inverse_transform 改善重建,可以在 KernelPCA 中調整 alpha,即在訓練映射期間控制對訓練資料依賴性的正規化項。
腳本總執行時間:(0 分鐘 0.614 秒)
相關範例