註記
前往結尾以下載完整的範例程式碼。或透過 JupyterLite 或 Binder 在您的瀏覽器中執行此範例
使用機率 PCA 和因子分析 (FA) 進行模型選擇#
機率 PCA 和因子分析是機率模型。結果是新資料的可能性可用於模型選擇和共變異數估計。這裡我們比較 PCA 和 FA,在低秩資料上進行交叉驗證,這些資料受到均方差雜訊(每個特徵的雜訊變異數相同)或異方差雜訊(每個特徵的雜訊變異數不同)的破壞。在第二步中,我們將模型可能性與從收縮共變異數估計器獲得的可能性進行比較。
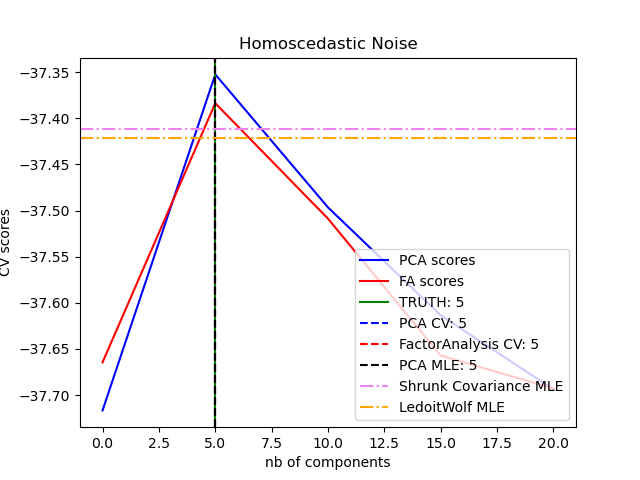
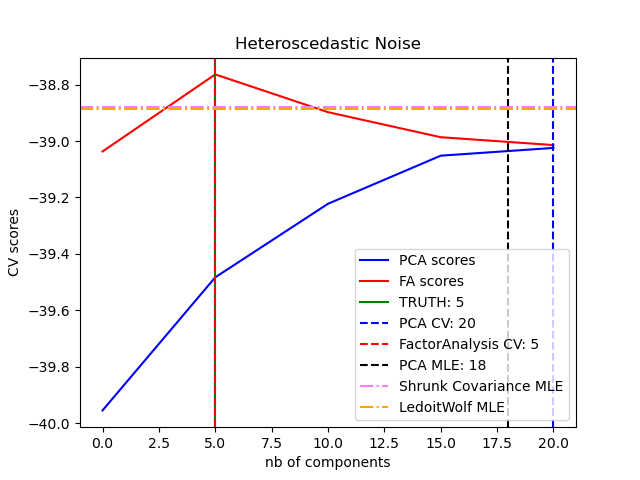
可以觀察到,在同方差雜訊的情況下,FA 和 PCA 都成功恢復了低秩子空間的大小。在這種情況下,PCA 的可能性高於 FA。然而,當存在異方差雜訊時,PCA 會失敗並高估秩。在適當的情況下(選擇成分數量),低秩模型的保留資料比收縮模型的可能性更高。
還比較了 Thomas P. Minka 在 NIPS 2000: 598-604 中的論文《自動選擇 PCA 維度》中提出的自動估計方法。
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
建立資料#
import numpy as np
from scipy import linalg
n_samples, n_features, rank = 500, 25, 5
sigma = 1.0
rng = np.random.RandomState(42)
U, _, _ = linalg.svd(rng.randn(n_features, n_features))
X = np.dot(rng.randn(n_samples, rank), U[:, :rank].T)
# Adding homoscedastic noise
X_homo = X + sigma * rng.randn(n_samples, n_features)
# Adding heteroscedastic noise
sigmas = sigma * rng.rand(n_features) + sigma / 2.0
X_hetero = X + rng.randn(n_samples, n_features) * sigmas
擬合模型#
import matplotlib.pyplot as plt
from sklearn.covariance import LedoitWolf, ShrunkCovariance
from sklearn.decomposition import PCA, FactorAnalysis
from sklearn.model_selection import GridSearchCV, cross_val_score
n_components = np.arange(0, n_features, 5) # options for n_components
def compute_scores(X):
pca = PCA(svd_solver="full")
fa = FactorAnalysis()
pca_scores, fa_scores = [], []
for n in n_components:
pca.n_components = n
fa.n_components = n
pca_scores.append(np.mean(cross_val_score(pca, X)))
fa_scores.append(np.mean(cross_val_score(fa, X)))
return pca_scores, fa_scores
def shrunk_cov_score(X):
shrinkages = np.logspace(-2, 0, 30)
cv = GridSearchCV(ShrunkCovariance(), {"shrinkage": shrinkages})
return np.mean(cross_val_score(cv.fit(X).best_estimator_, X))
def lw_score(X):
return np.mean(cross_val_score(LedoitWolf(), X))
for X, title in [(X_homo, "Homoscedastic Noise"), (X_hetero, "Heteroscedastic Noise")]:
pca_scores, fa_scores = compute_scores(X)
n_components_pca = n_components[np.argmax(pca_scores)]
n_components_fa = n_components[np.argmax(fa_scores)]
pca = PCA(svd_solver="full", n_components="mle")
pca.fit(X)
n_components_pca_mle = pca.n_components_
print("best n_components by PCA CV = %d" % n_components_pca)
print("best n_components by FactorAnalysis CV = %d" % n_components_fa)
print("best n_components by PCA MLE = %d" % n_components_pca_mle)
plt.figure()
plt.plot(n_components, pca_scores, "b", label="PCA scores")
plt.plot(n_components, fa_scores, "r", label="FA scores")
plt.axvline(rank, color="g", label="TRUTH: %d" % rank, linestyle="-")
plt.axvline(
n_components_pca,
color="b",
label="PCA CV: %d" % n_components_pca,
linestyle="--",
)
plt.axvline(
n_components_fa,
color="r",
label="FactorAnalysis CV: %d" % n_components_fa,
linestyle="--",
)
plt.axvline(
n_components_pca_mle,
color="k",
label="PCA MLE: %d" % n_components_pca_mle,
linestyle="--",
)
# compare with other covariance estimators
plt.axhline(
shrunk_cov_score(X),
color="violet",
label="Shrunk Covariance MLE",
linestyle="-.",
)
plt.axhline(
lw_score(X),
color="orange",
label="LedoitWolf MLE" % n_components_pca_mle,
linestyle="-.",
)
plt.xlabel("nb of components")
plt.ylabel("CV scores")
plt.legend(loc="lower right")
plt.title(title)
plt.show()
best n_components by PCA CV = 5
best n_components by FactorAnalysis CV = 5
best n_components by PCA MLE = 5
best n_components by PCA CV = 20
best n_components by FactorAnalysis CV = 5
best n_components by PCA MLE = 18
腳本的總執行時間:(0 分鐘 3.135 秒)
相關範例