注意
前往結尾以下載完整的範例程式碼。或透過 JupyterLite 或 Binder 在您的瀏覽器中執行此範例
穩健共變異數估計和馬氏距離的相關性#
此範例顯示高斯分佈資料的馬氏距離的共變異數估計。
對於高斯分佈資料,可以使用馬氏距離計算觀測值 \(x_i\) 到分佈模式的距離
其中 \(\mu\) 和 \(\Sigma\) 是基礎高斯分佈的位置和共變異數。
實際上,\(\mu\) 和 \(\Sigma\) 由某些估計值取代。標準共變異數最大概似估計 (MLE) 對資料集中離群值的存在非常敏感,因此,下游馬氏距離也是如此。最好使用穩健的共變異數估計器,以確保估計值能夠抵抗資料集中的「錯誤」觀測值,並且計算出的馬氏距離能夠準確反映觀測值的真實組織。
最小共變異數行列式估計器 (MCD) 是一種穩健的高崩潰點(即它可用於估計高污染資料集的共變異數矩陣,最多可達 \(\frac{n_\text{samples}-n_\text{features}-1}{2}\) 個離群值)共變異數估計器。MCD 背後的概念是找到 \(\frac{n_\text{samples}+n_\text{features}+1}{2}\) 個觀測值,這些觀測值的經驗共變異數具有最小行列式,從而產生「純」觀測值子集,從中計算位置和共變異數的標準估計值。MCD 由 P.J.Rousseuw 在 [1] 中提出。
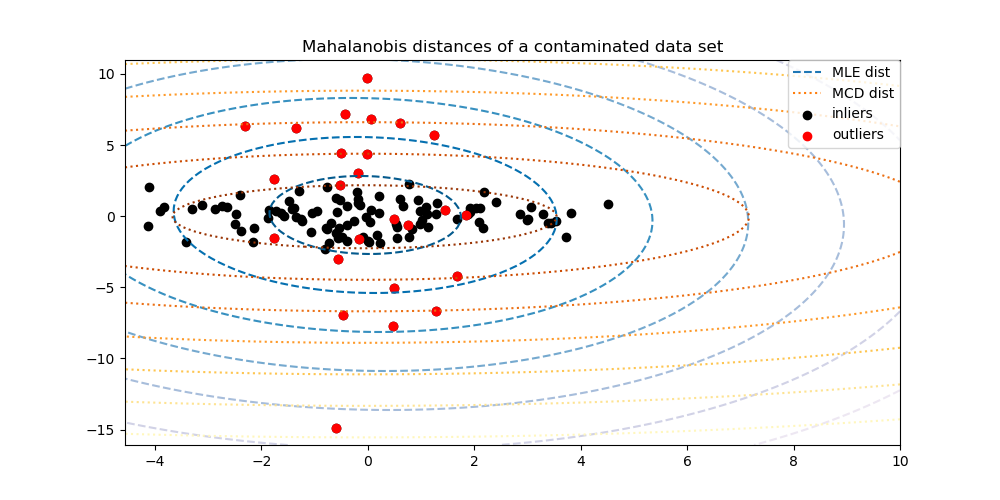
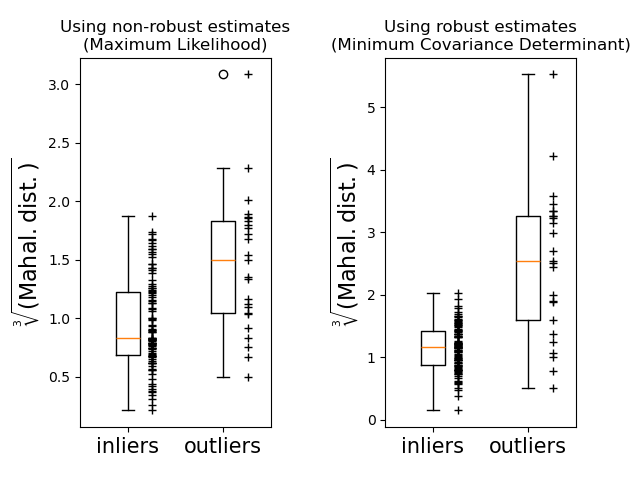
此範例說明馬氏距離如何受到離群資料的影響。當使用基於標準共變異數 MLE 的馬氏距離時,從污染分佈中繪製的觀測值與來自真實高斯分佈的觀測值無法區分。使用基於 MCD 的馬氏距離,兩個群體變得可以區分。相關應用包括離群值檢測、觀測值排名和分群。
注意
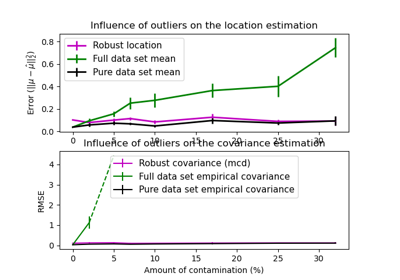
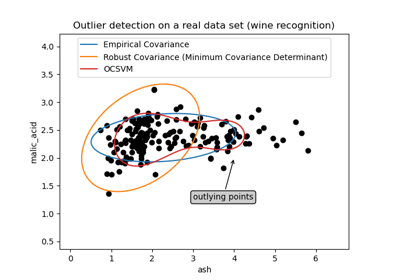
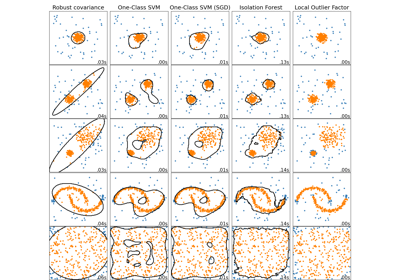
另請參閱 穩健與經驗共變異數估計
參考文獻
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
產生資料#
首先,我們產生一個包含 125 個樣本和 2 個特徵的資料集。兩個特徵都是高斯分佈,平均值為 0,但特徵 1 的標準差等於 2,而特徵 2 的標準差等於 1。接下來,25 個樣本被高斯離群值樣本取代,其中特徵 1 的標準差等於 1,而特徵 2 的標準差等於 7。
import numpy as np
# for consistent results
np.random.seed(7)
n_samples = 125
n_outliers = 25
n_features = 2
# generate Gaussian data of shape (125, 2)
gen_cov = np.eye(n_features)
gen_cov[0, 0] = 2.0
X = np.dot(np.random.randn(n_samples, n_features), gen_cov)
# add some outliers
outliers_cov = np.eye(n_features)
outliers_cov[np.arange(1, n_features), np.arange(1, n_features)] = 7.0
X[-n_outliers:] = np.dot(np.random.randn(n_outliers, n_features), outliers_cov)
結果比較#
在下面,我們將基於 MCD 和 MLE 的共變異數估計器擬合到我們的資料,並列印估計的共變異數矩陣。請注意,基於 MLE 的估計器(7.5)的特徵 2 的估計變異數比 MCD 穩健估計器(1.2)的估計變異數高得多。這表明,基於 MCD 的穩健估計器更能抵抗離群值樣本,這些樣本的特徵 2 具有更大的變異數。
import matplotlib.pyplot as plt
from sklearn.covariance import EmpiricalCovariance, MinCovDet
# fit a MCD robust estimator to data
robust_cov = MinCovDet().fit(X)
# fit a MLE estimator to data
emp_cov = EmpiricalCovariance().fit(X)
print(
"Estimated covariance matrix:\nMCD (Robust):\n{}\nMLE:\n{}".format(
robust_cov.covariance_, emp_cov.covariance_
)
)
Estimated covariance matrix:
MCD (Robust):
[[ 3.26253567e+00 -3.06695631e-03]
[-3.06695631e-03 1.22747343e+00]]
MLE:
[[ 3.23773583 -0.24640578]
[-0.24640578 7.51963999]]
為了更好地視覺化差異,我們繪製由兩種方法計算出的馬氏距離的等高線。請注意,基於穩健 MCD 的馬氏距離更適合內點黑點,而基於 MLE 的距離則更容易受到離群值紅點的影響。
import matplotlib.lines as mlines
fig, ax = plt.subplots(figsize=(10, 5))
# Plot data set
inlier_plot = ax.scatter(X[:, 0], X[:, 1], color="black", label="inliers")
outlier_plot = ax.scatter(
X[:, 0][-n_outliers:], X[:, 1][-n_outliers:], color="red", label="outliers"
)
ax.set_xlim(ax.get_xlim()[0], 10.0)
ax.set_title("Mahalanobis distances of a contaminated data set")
# Create meshgrid of feature 1 and feature 2 values
xx, yy = np.meshgrid(
np.linspace(plt.xlim()[0], plt.xlim()[1], 100),
np.linspace(plt.ylim()[0], plt.ylim()[1], 100),
)
zz = np.c_[xx.ravel(), yy.ravel()]
# Calculate the MLE based Mahalanobis distances of the meshgrid
mahal_emp_cov = emp_cov.mahalanobis(zz)
mahal_emp_cov = mahal_emp_cov.reshape(xx.shape)
emp_cov_contour = plt.contour(
xx, yy, np.sqrt(mahal_emp_cov), cmap=plt.cm.PuBu_r, linestyles="dashed"
)
# Calculate the MCD based Mahalanobis distances
mahal_robust_cov = robust_cov.mahalanobis(zz)
mahal_robust_cov = mahal_robust_cov.reshape(xx.shape)
robust_contour = ax.contour(
xx, yy, np.sqrt(mahal_robust_cov), cmap=plt.cm.YlOrBr_r, linestyles="dotted"
)
# Add legend
ax.legend(
[
mlines.Line2D([], [], color="tab:blue", linestyle="dashed"),
mlines.Line2D([], [], color="tab:orange", linestyle="dotted"),
inlier_plot,
outlier_plot,
],
["MLE dist", "MCD dist", "inliers", "outliers"],
loc="upper right",
borderaxespad=0,
)
plt.show()
最後,我們強調基於 MCD 的馬氏距離區分離群值的能力。我們取馬氏距離的立方根,產生近似常態分佈(如 Wilson 和 Hilferty [2] 所建議),然後使用盒狀圖繪製內點和離群值樣本的值。對於基於穩健 MCD 的馬氏距離,離群值樣本的分佈與內點樣本的分佈更為分離。
fig, (ax1, ax2) = plt.subplots(1, 2)
plt.subplots_adjust(wspace=0.6)
# Calculate cubic root of MLE Mahalanobis distances for samples
emp_mahal = emp_cov.mahalanobis(X - np.mean(X, 0)) ** (0.33)
# Plot boxplots
ax1.boxplot([emp_mahal[:-n_outliers], emp_mahal[-n_outliers:]], widths=0.25)
# Plot individual samples
ax1.plot(
np.full(n_samples - n_outliers, 1.26),
emp_mahal[:-n_outliers],
"+k",
markeredgewidth=1,
)
ax1.plot(np.full(n_outliers, 2.26), emp_mahal[-n_outliers:], "+k", markeredgewidth=1)
ax1.axes.set_xticklabels(("inliers", "outliers"), size=15)
ax1.set_ylabel(r"$\sqrt[3]{\rm{(Mahal. dist.)}}$", size=16)
ax1.set_title("Using non-robust estimates\n(Maximum Likelihood)")
# Calculate cubic root of MCD Mahalanobis distances for samples
robust_mahal = robust_cov.mahalanobis(X - robust_cov.location_) ** (0.33)
# Plot boxplots
ax2.boxplot([robust_mahal[:-n_outliers], robust_mahal[-n_outliers:]], widths=0.25)
# Plot individual samples
ax2.plot(
np.full(n_samples - n_outliers, 1.26),
robust_mahal[:-n_outliers],
"+k",
markeredgewidth=1,
)
ax2.plot(np.full(n_outliers, 2.26), robust_mahal[-n_outliers:], "+k", markeredgewidth=1)
ax2.axes.set_xticklabels(("inliers", "outliers"), size=15)
ax2.set_ylabel(r"$\sqrt[3]{\rm{(Mahal. dist.)}}$", size=16)
ax2.set_title("Using robust estimates\n(Minimum Covariance Determinant)")
plt.show()
腳本的總執行時間: (0 分鐘 0.277 秒)
相關範例