注意
前往結尾以下載完整的範例程式碼。或透過 JupyterLite 或 Binder 在您的瀏覽器中執行此範例
具有多重共線性或相關特徵的排列重要性#
在此範例中,我們計算訓練過的 RandomForestClassifier 的特徵的 permutation_importance,使用 乳癌威斯康辛 (診斷) 資料集。該模型在測試資料集上可以輕鬆獲得約 97% 的準確度。由於此資料集包含多重共線性特徵,因此排列重要性顯示沒有任何特徵是重要的,這與高測試準確度相矛盾。
我們示範了一種處理多重共線性的可能方法,該方法包括對特徵的 Spearman 秩次相關性進行層次聚類、選擇閾值,並從每個群集中保留單一特徵。
注意
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
乳癌資料上的隨機森林特徵重要性#
首先,我們定義一個函數來簡化繪圖
import matplotlib
from sklearn.inspection import permutation_importance
from sklearn.utils.fixes import parse_version
def plot_permutation_importance(clf, X, y, ax):
result = permutation_importance(clf, X, y, n_repeats=10, random_state=42, n_jobs=2)
perm_sorted_idx = result.importances_mean.argsort()
# `labels` argument in boxplot is deprecated in matplotlib 3.9 and has been
# renamed to `tick_labels`. The following code handles this, but as a
# scikit-learn user you probably can write simpler code by using `labels=...`
# (matplotlib < 3.9) or `tick_labels=...` (matplotlib >= 3.9).
tick_labels_parameter_name = (
"tick_labels"
if parse_version(matplotlib.__version__) >= parse_version("3.9")
else "labels"
)
tick_labels_dict = {tick_labels_parameter_name: X.columns[perm_sorted_idx]}
ax.boxplot(result.importances[perm_sorted_idx].T, vert=False, **tick_labels_dict)
ax.axvline(x=0, color="k", linestyle="--")
return ax
然後,我們在 乳癌威斯康辛 (診斷) 資料集上訓練 RandomForestClassifier,並評估其在測試集上的準確度
from sklearn.datasets import load_breast_cancer
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import train_test_split
X, y = load_breast_cancer(return_X_y=True, as_frame=True)
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=42)
clf = RandomForestClassifier(n_estimators=100, random_state=42)
clf.fit(X_train, y_train)
print(f"Baseline accuracy on test data: {clf.score(X_test, y_test):.2}")
Baseline accuracy on test data: 0.97
接下來,我們繪製基於樹的特徵重要性和排列重要性。排列重要性是在訓練集上計算的,以顯示模型在訓練期間依賴每個特徵的程度。
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
mdi_importances = pd.Series(clf.feature_importances_, index=X_train.columns)
tree_importance_sorted_idx = np.argsort(clf.feature_importances_)
fig, (ax1, ax2) = plt.subplots(1, 2, figsize=(12, 8))
mdi_importances.sort_values().plot.barh(ax=ax1)
ax1.set_xlabel("Gini importance")
plot_permutation_importance(clf, X_train, y_train, ax2)
ax2.set_xlabel("Decrease in accuracy score")
fig.suptitle(
"Impurity-based vs. permutation importances on multicollinear features (train set)"
)
_ = fig.tight_layout()
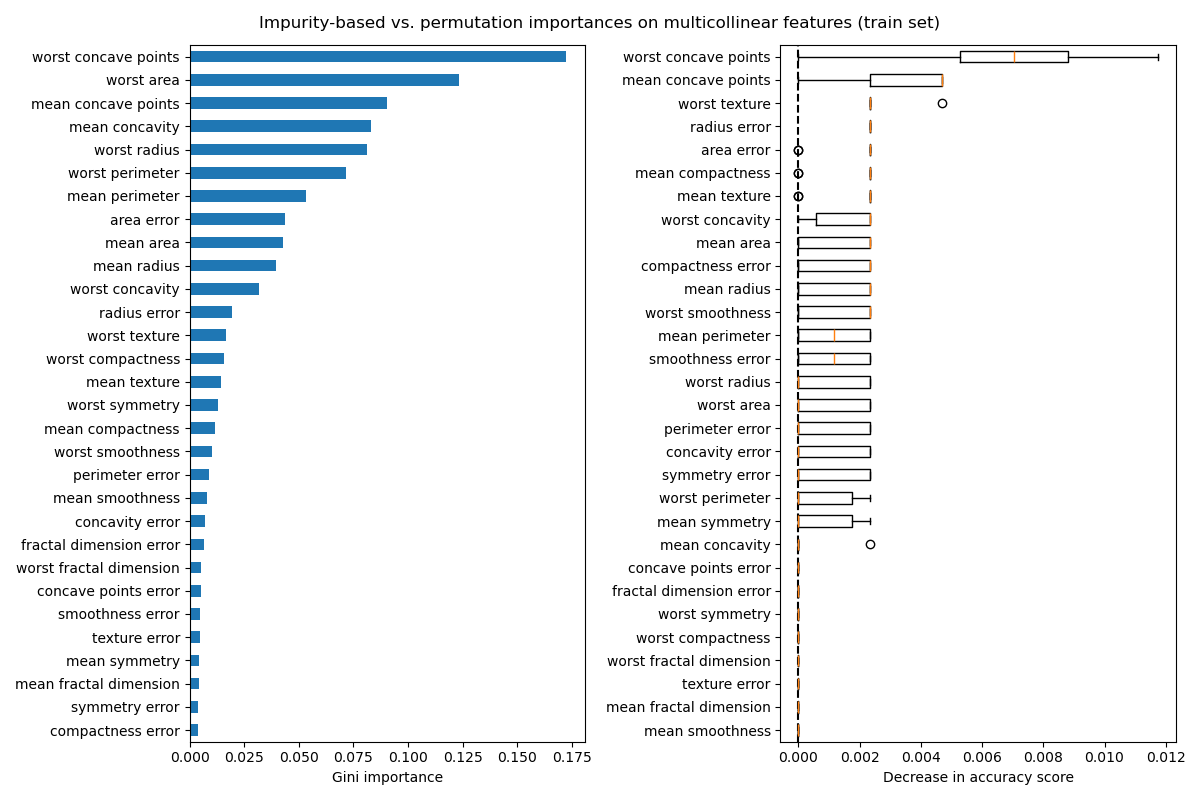
左側的圖顯示了模型的 Gini 重要性。由於 RandomForestClassifier 的 scikit-learn 實作在每次分割時使用 \(\sqrt{n_\text{features}}\) 個特徵的隨機子集,因此它能夠稀釋任何單一相關特徵的優勢。因此,個別特徵的重要性可能會更均勻地分佈在相關特徵之間。由於這些特徵具有較大的基數且分類器沒有過擬合,因此我們可以相對信任這些值。
右圖上的排列重要性顯示,排列一個特徵最多會使準確度下降 0.012,這表明沒有任何特徵是重要的。這與計算為基準的高測試準確度相矛盾:某些特徵一定很重要。
同樣地,在測試集上計算的準確度分數變化似乎是由於機率驅動的
fig, ax = plt.subplots(figsize=(7, 6))
plot_permutation_importance(clf, X_test, y_test, ax)
ax.set_title("Permutation Importances on multicollinear features\n(test set)")
ax.set_xlabel("Decrease in accuracy score")
_ = ax.figure.tight_layout()
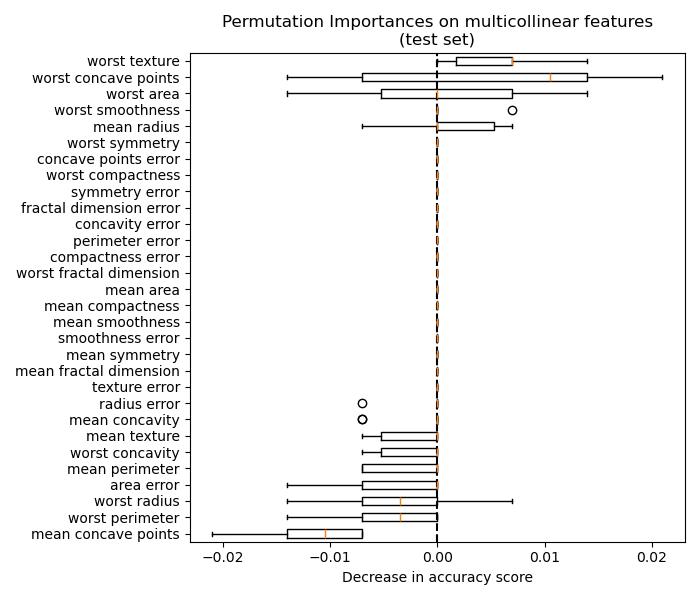
儘管如此,在存在相關特徵的情況下,仍然可以計算出有意義的排列重要性,如下節所示。
處理多重共線性特徵#
當特徵共線時,排列一個特徵對模型的效能影響不大,因為它可以從相關特徵獲得相同的資訊。請注意,並非所有預測模型都是如此,這取決於它們的底層實作。
處理多重共線性特徵的一種方法是對 Spearman 秩次相關性進行層次聚類、選擇閾值,並從每個群集中保留單一特徵。首先,我們繪製相關特徵的熱圖
from scipy.cluster import hierarchy
from scipy.spatial.distance import squareform
from scipy.stats import spearmanr
fig, (ax1, ax2) = plt.subplots(1, 2, figsize=(12, 8))
corr = spearmanr(X).correlation
# Ensure the correlation matrix is symmetric
corr = (corr + corr.T) / 2
np.fill_diagonal(corr, 1)
# We convert the correlation matrix to a distance matrix before performing
# hierarchical clustering using Ward's linkage.
distance_matrix = 1 - np.abs(corr)
dist_linkage = hierarchy.ward(squareform(distance_matrix))
dendro = hierarchy.dendrogram(
dist_linkage, labels=X.columns.to_list(), ax=ax1, leaf_rotation=90
)
dendro_idx = np.arange(0, len(dendro["ivl"]))
ax2.imshow(corr[dendro["leaves"], :][:, dendro["leaves"]])
ax2.set_xticks(dendro_idx)
ax2.set_yticks(dendro_idx)
ax2.set_xticklabels(dendro["ivl"], rotation="vertical")
ax2.set_yticklabels(dendro["ivl"])
_ = fig.tight_layout()
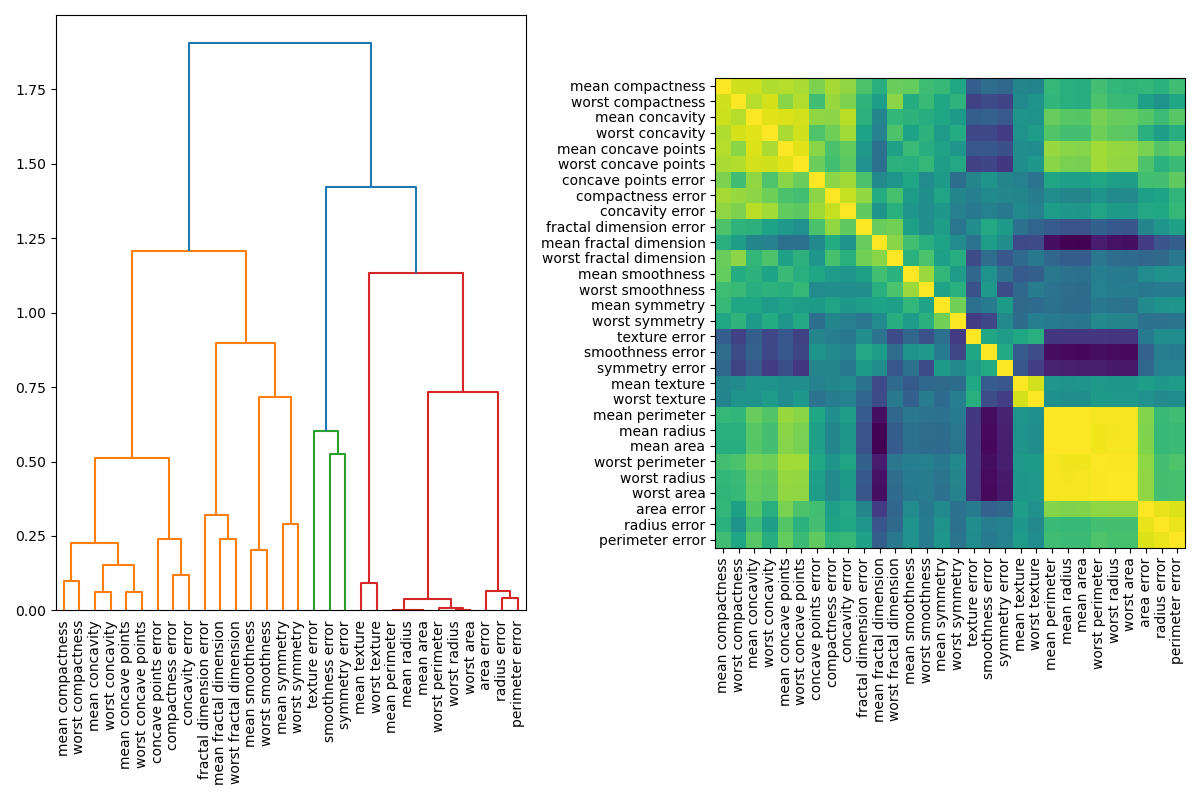
接下來,我們透過目視檢查樹狀圖手動選擇一個閾值,將我們的特徵分組到群集中,並選擇每個群集中的一個特徵來保留,從我們的資料集中選擇這些特徵,並訓練新的隨機森林。與在完整資料集上訓練的隨機森林相比,新的隨機森林的測試準確度沒有太大變化。
from collections import defaultdict
cluster_ids = hierarchy.fcluster(dist_linkage, 1, criterion="distance")
cluster_id_to_feature_ids = defaultdict(list)
for idx, cluster_id in enumerate(cluster_ids):
cluster_id_to_feature_ids[cluster_id].append(idx)
selected_features = [v[0] for v in cluster_id_to_feature_ids.values()]
selected_features_names = X.columns[selected_features]
X_train_sel = X_train[selected_features_names]
X_test_sel = X_test[selected_features_names]
clf_sel = RandomForestClassifier(n_estimators=100, random_state=42)
clf_sel.fit(X_train_sel, y_train)
print(
"Baseline accuracy on test data with features removed:"
f" {clf_sel.score(X_test_sel, y_test):.2}"
)
Baseline accuracy on test data with features removed: 0.97
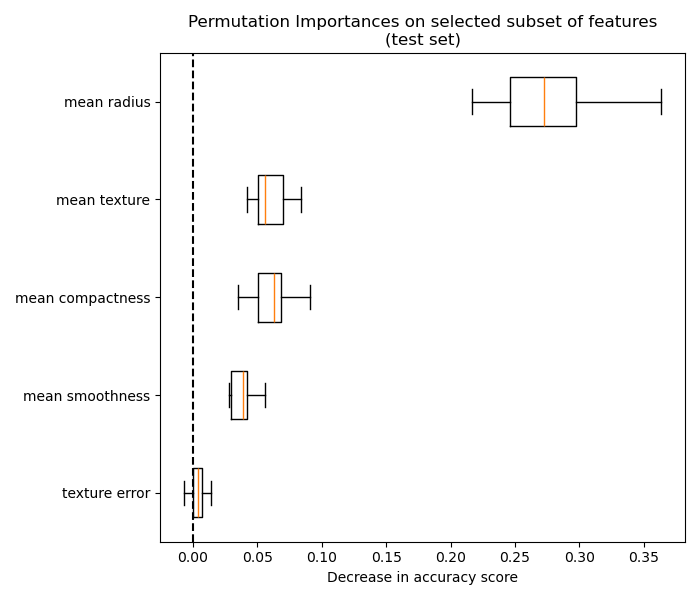
我們最終可以探索所選特徵子集的排列重要性
fig, ax = plt.subplots(figsize=(7, 6))
plot_permutation_importance(clf_sel, X_test_sel, y_test, ax)
ax.set_title("Permutation Importances on selected subset of features\n(test set)")
ax.set_xlabel("Decrease in accuracy score")
ax.figure.tight_layout()
plt.show()
腳本的總執行時間: (0 分鐘 6.532 秒)
相關範例