注意
前往結尾下載完整的範例程式碼。或透過 JupyterLite 或 Binder 在您的瀏覽器中執行此範例
HDBSCAN 群集演算法的演示#
在此演示中,我們將從推廣 cluster.DBSCAN 演算法的角度來看看 cluster.HDBSCAN。我們將在特定數據集上比較這兩種演算法。最後,我們將評估 HDBSCAN 對某些超參數的敏感度。
我們先定義一些方便的實用函式。
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
import matplotlib.pyplot as plt
import numpy as np
from sklearn.cluster import DBSCAN, HDBSCAN
from sklearn.datasets import make_blobs
def plot(X, labels, probabilities=None, parameters=None, ground_truth=False, ax=None):
if ax is None:
_, ax = plt.subplots(figsize=(10, 4))
labels = labels if labels is not None else np.ones(X.shape[0])
probabilities = probabilities if probabilities is not None else np.ones(X.shape[0])
# Black removed and is used for noise instead.
unique_labels = set(labels)
colors = [plt.cm.Spectral(each) for each in np.linspace(0, 1, len(unique_labels))]
# The probability of a point belonging to its labeled cluster determines
# the size of its marker
proba_map = {idx: probabilities[idx] for idx in range(len(labels))}
for k, col in zip(unique_labels, colors):
if k == -1:
# Black used for noise.
col = [0, 0, 0, 1]
class_index = np.where(labels == k)[0]
for ci in class_index:
ax.plot(
X[ci, 0],
X[ci, 1],
"x" if k == -1 else "o",
markerfacecolor=tuple(col),
markeredgecolor="k",
markersize=4 if k == -1 else 1 + 5 * proba_map[ci],
)
n_clusters_ = len(set(labels)) - (1 if -1 in labels else 0)
preamble = "True" if ground_truth else "Estimated"
title = f"{preamble} number of clusters: {n_clusters_}"
if parameters is not None:
parameters_str = ", ".join(f"{k}={v}" for k, v in parameters.items())
title += f" | {parameters_str}"
ax.set_title(title)
plt.tight_layout()
產生樣本數據#
HDBSCAN 相較於 DBSCAN 的最大優勢之一是其開箱即用的穩健性。它在異質數據混合物上尤其出色。與 DBSCAN 一樣,它可以模擬任意形狀和分佈,但與 DBSCAN 不同的是,它不需要指定任意且敏感的 eps 超參數。
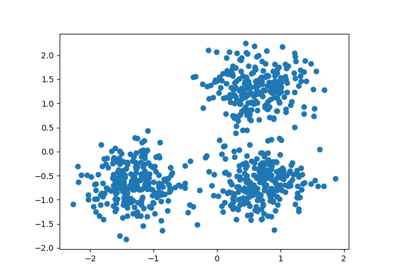
例如,以下我們從三個二維和各向同性高斯分佈的混合中產生一個數據集。
centers = [[1, 1], [-1, -1], [1.5, -1.5]]
X, labels_true = make_blobs(
n_samples=750, centers=centers, cluster_std=[0.4, 0.1, 0.75], random_state=0
)
plot(X, labels=labels_true, ground_truth=True)
尺度不變性#
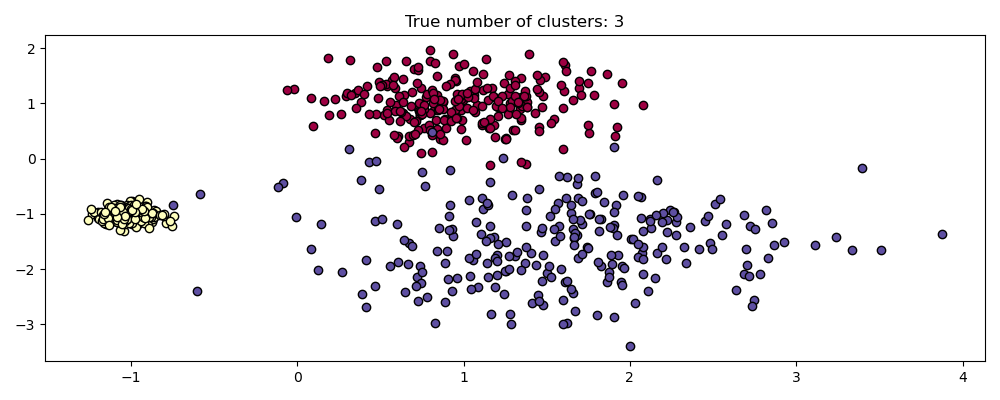
值得記住的是,雖然 DBSCAN 為 eps 參數提供了預設值,但它幾乎沒有適當的預設值,必須針對使用的特定數據集進行調整。
作為一個簡單的演示,考慮針對一個數據集調整的 eps 值的群集,以及使用相同值但應用於數據集重新縮放版本獲得的群集。
fig, axes = plt.subplots(3, 1, figsize=(10, 12))
dbs = DBSCAN(eps=0.3)
for idx, scale in enumerate([1, 0.5, 3]):
dbs.fit(X * scale)
plot(X * scale, dbs.labels_, parameters={"scale": scale, "eps": 0.3}, ax=axes[idx])
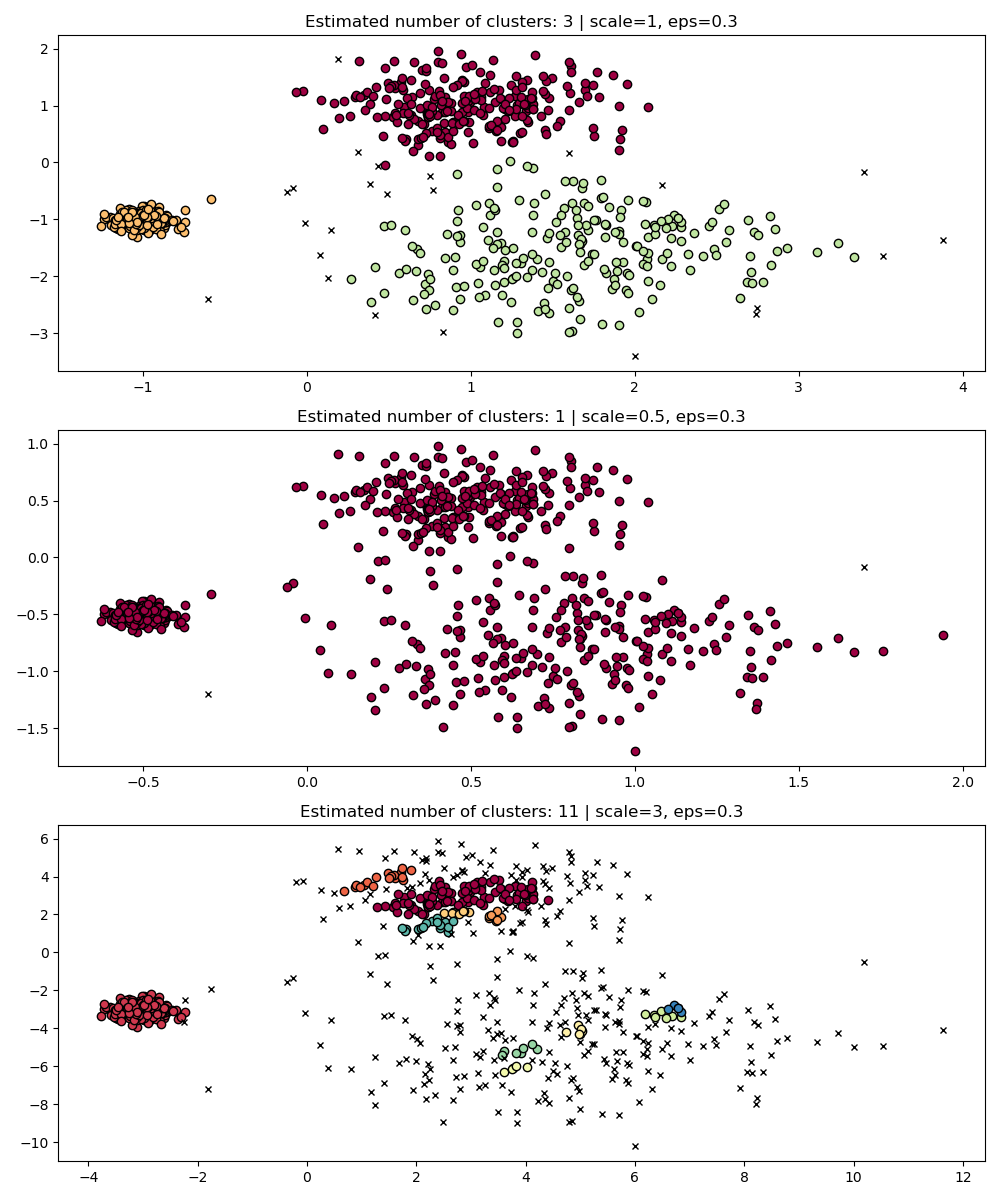
實際上,為了保持相同的結果,我們必須將 eps 按相同的因子縮放。
fig, axis = plt.subplots(1, 1, figsize=(12, 5))
dbs = DBSCAN(eps=0.9).fit(3 * X)
plot(3 * X, dbs.labels_, parameters={"scale": 3, "eps": 0.9}, ax=axis)
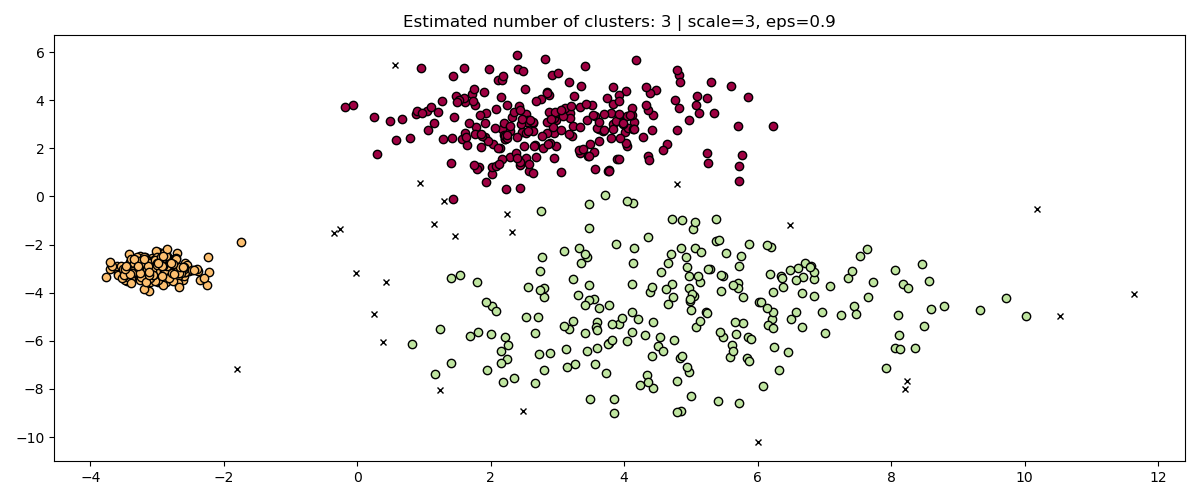
雖然標準化數據(例如,使用 sklearn.preprocessing.StandardScaler)有助於減輕這個問題,但必須非常小心地為 eps 選擇適當的值。
HDBSCAN 在這方面更穩健:HDBSCAN 可以看作是對 eps 所有可能的值進行群集,並從所有可能的群集中提取最佳群集(請參閱使用者指南)。一個直接的優勢是 HDBSCAN 具有尺度不變性。
fig, axes = plt.subplots(3, 1, figsize=(10, 12))
hdb = HDBSCAN()
for idx, scale in enumerate([1, 0.5, 3]):
hdb.fit(X * scale)
plot(
X * scale,
hdb.labels_,
hdb.probabilities_,
ax=axes[idx],
parameters={"scale": scale},
)
多尺度群集#
不過,HDBSCAN 不僅僅是尺度不變的 – 它能夠進行多尺度群集,這考慮到具有不同密度的群集。傳統的 DBSCAN 假設任何潛在的群集在密度上都是同質的。HDBSCAN 不受這些約束。為了演示這一點,我們考慮以下數據集
centers = [[-0.85, -0.85], [-0.85, 0.85], [3, 3], [3, -3]]
X, labels_true = make_blobs(
n_samples=750, centers=centers, cluster_std=[0.2, 0.35, 1.35, 1.35], random_state=0
)
plot(X, labels=labels_true, ground_truth=True)
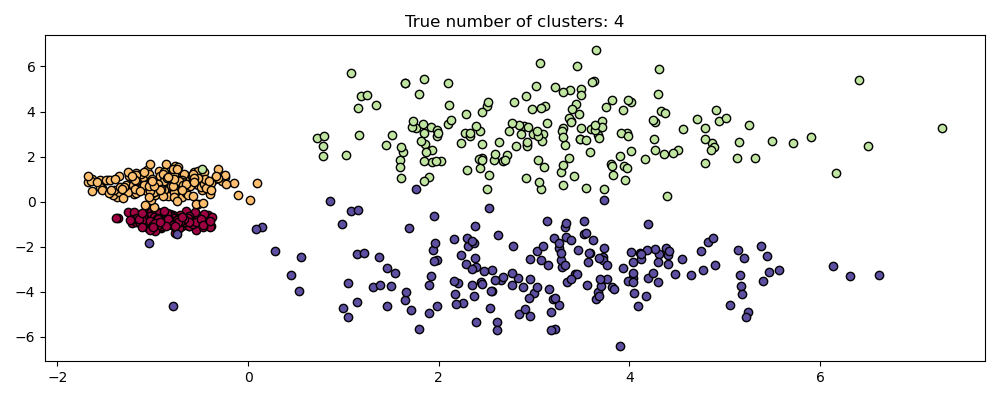
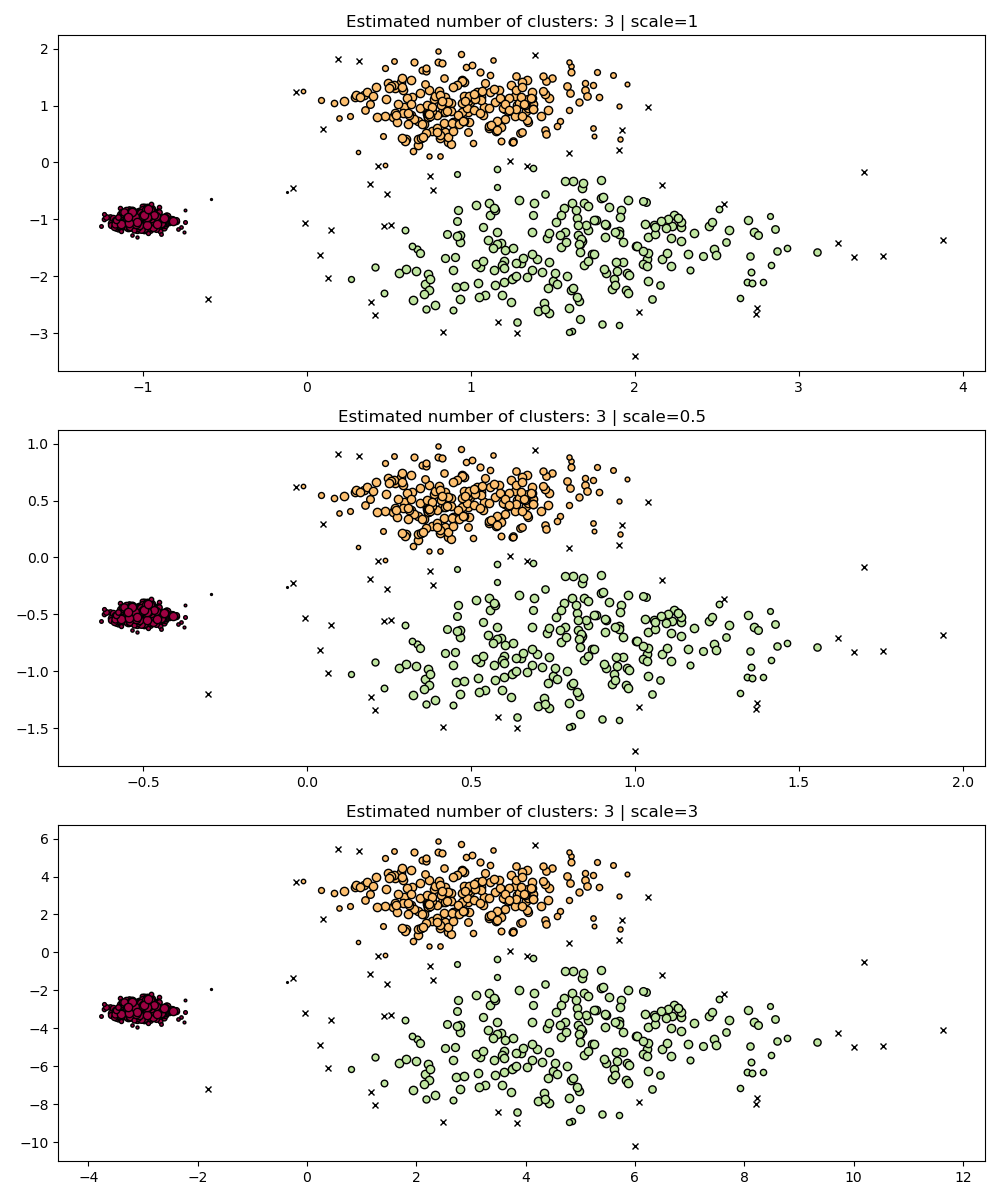
由於密度和空間分隔的不同,此數據集對於 DBSCAN 來說更困難
如果
eps太大,那麼我們可能會錯誤地將兩個密集群集群集為一個,因為它們的相互可達性將擴展群集。如果
eps太小,那麼我們可能會將較稀疏的群集分割成許多錯誤的群集。
更不用說這需要手動調整 eps 的選擇,直到我們找到一個我們覺得舒適的權衡。
fig, axes = plt.subplots(2, 1, figsize=(10, 8))
params = {"eps": 0.7}
dbs = DBSCAN(**params).fit(X)
plot(X, dbs.labels_, parameters=params, ax=axes[0])
params = {"eps": 0.3}
dbs = DBSCAN(**params).fit(X)
plot(X, dbs.labels_, parameters=params, ax=axes[1])
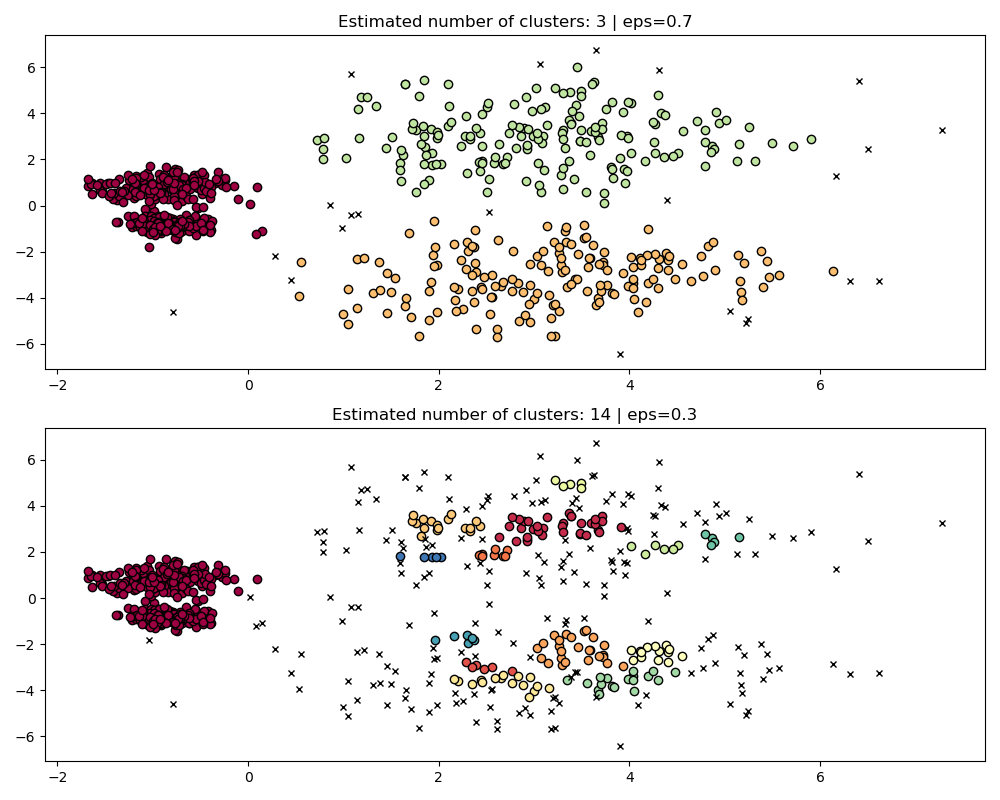
為了正確群集兩個密集群集,我們需要一個較小的 epsilon 值,但是當 eps=0.3 時,我們已經在分割稀疏群集,當我們減少 epsilon 時,情況只會變得更嚴重。事實上,DBSCAN 似乎無法同時分離兩個密集群集,同時防止稀疏群集被分割。讓我們與 HDBSCAN 比較。
hdb = HDBSCAN().fit(X)
plot(X, hdb.labels_, hdb.probabilities_)
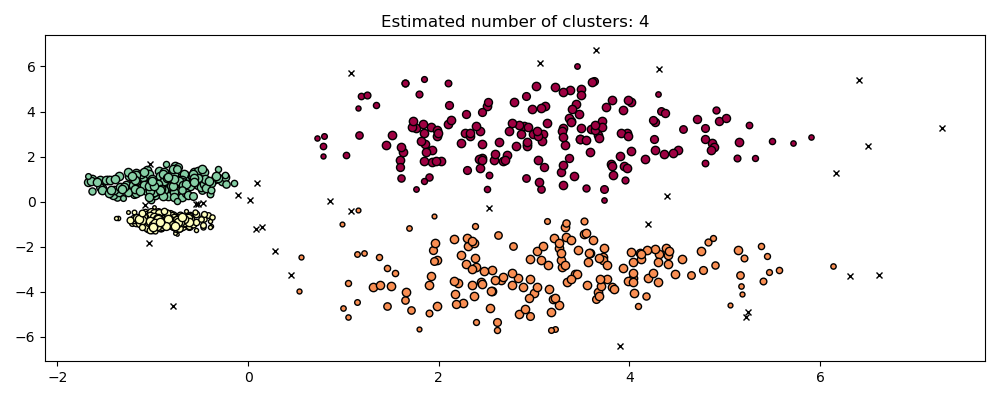
HDBSCAN 能夠適應數據集的多尺度結構,而無需進行參數調整。雖然任何足夠有趣的數據集都需要調整,但這種情況表明 HDBSCAN 可以在沒有用戶干預的情況下產生定性上更好的群集類別,而 DBSCAN 無法實現。
超參數穩健性#
最終,調整將是任何現實世界應用程式中的重要步驟,因此讓我們看看 HDBSCAN 的一些最重要的超參數。雖然 HDBSCAN 沒有 DBSCAN 的 eps 參數,但它仍然有一些超參數,例如 min_cluster_size 和 min_samples,它們會調整其關於密度的結果。不過,我們將看到,由於這些參數的明確含義有助於調整它們,HDBSCAN 相對來說對各種現實世界範例具有穩健性。
min_cluster_size#
min_cluster_size 是群組中要被視為群集的最小樣本數。
小於此大小的群集將被保留為雜訊。預設值為 5。此參數通常根據需要調整為更大的值。較小的值可能會導致較少的點被標記為雜訊的結果。但是,太小的值會導致錯誤的子群集被拾取和優先處理。較大的值往往對嘈雜的數據集(例如,具有顯著重疊的高方差群集)更穩健。
PARAM = ({"min_cluster_size": 5}, {"min_cluster_size": 3}, {"min_cluster_size": 25})
fig, axes = plt.subplots(3, 1, figsize=(10, 12))
for i, param in enumerate(PARAM):
hdb = HDBSCAN(**param).fit(X)
labels = hdb.labels_
plot(X, labels, hdb.probabilities_, param, ax=axes[i])
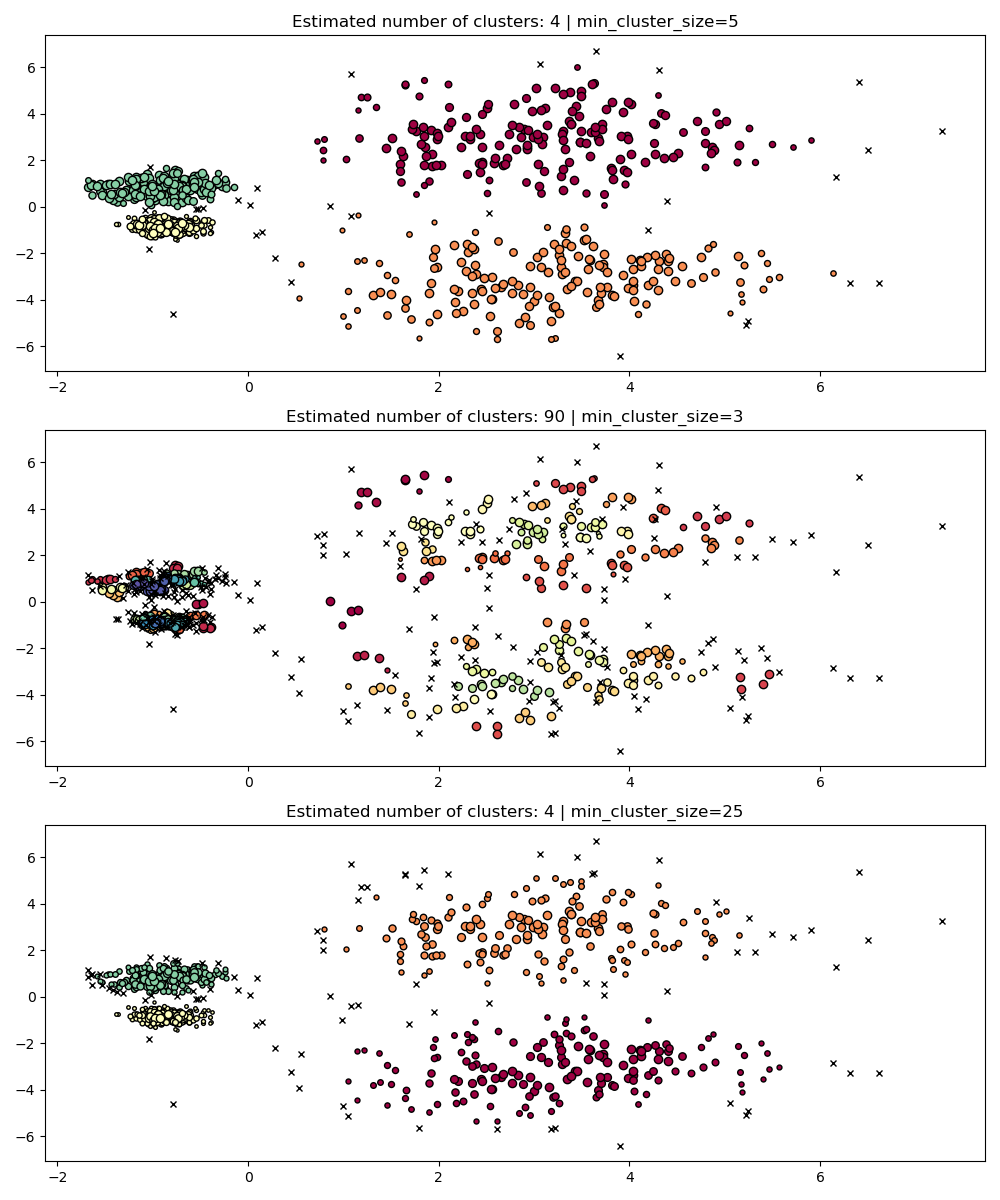
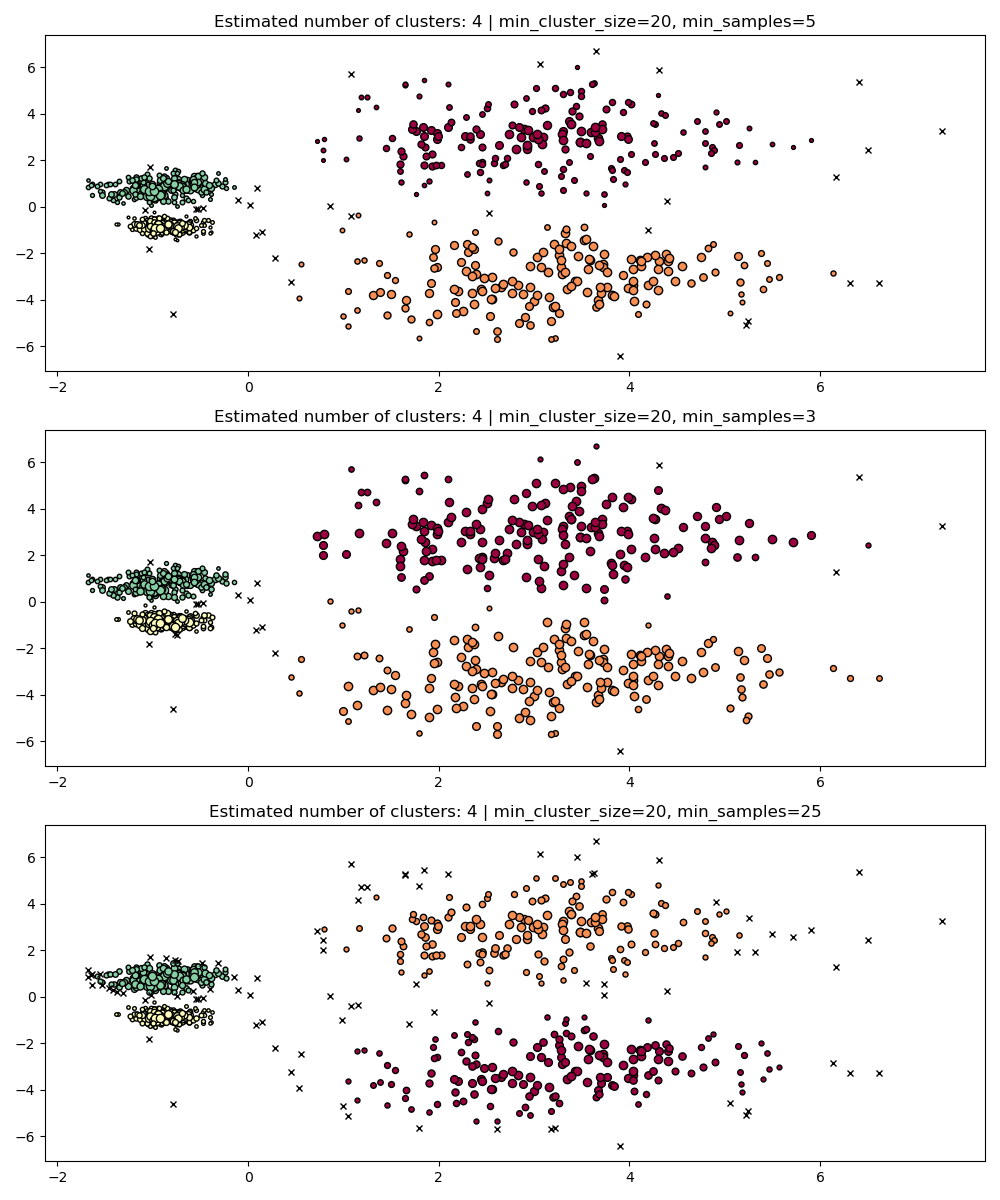
min_samples#
min_samples 是鄰域中要被視為核心點的樣本數,包括該點本身。min_samples 預設為 min_cluster_size。與 min_cluster_size 類似,min_samples 的較大值會增加模型對雜訊的穩健性,但有可能忽略或丟棄潛在有效但較小的群集。min_samples 最好在找到 min_cluster_size 的良好值後再進行調整。
PARAM = (
{"min_cluster_size": 20, "min_samples": 5},
{"min_cluster_size": 20, "min_samples": 3},
{"min_cluster_size": 20, "min_samples": 25},
)
fig, axes = plt.subplots(3, 1, figsize=(10, 12))
for i, param in enumerate(PARAM):
hdb = HDBSCAN(**param).fit(X)
labels = hdb.labels_
plot(X, labels, hdb.probabilities_, param, ax=axes[i])
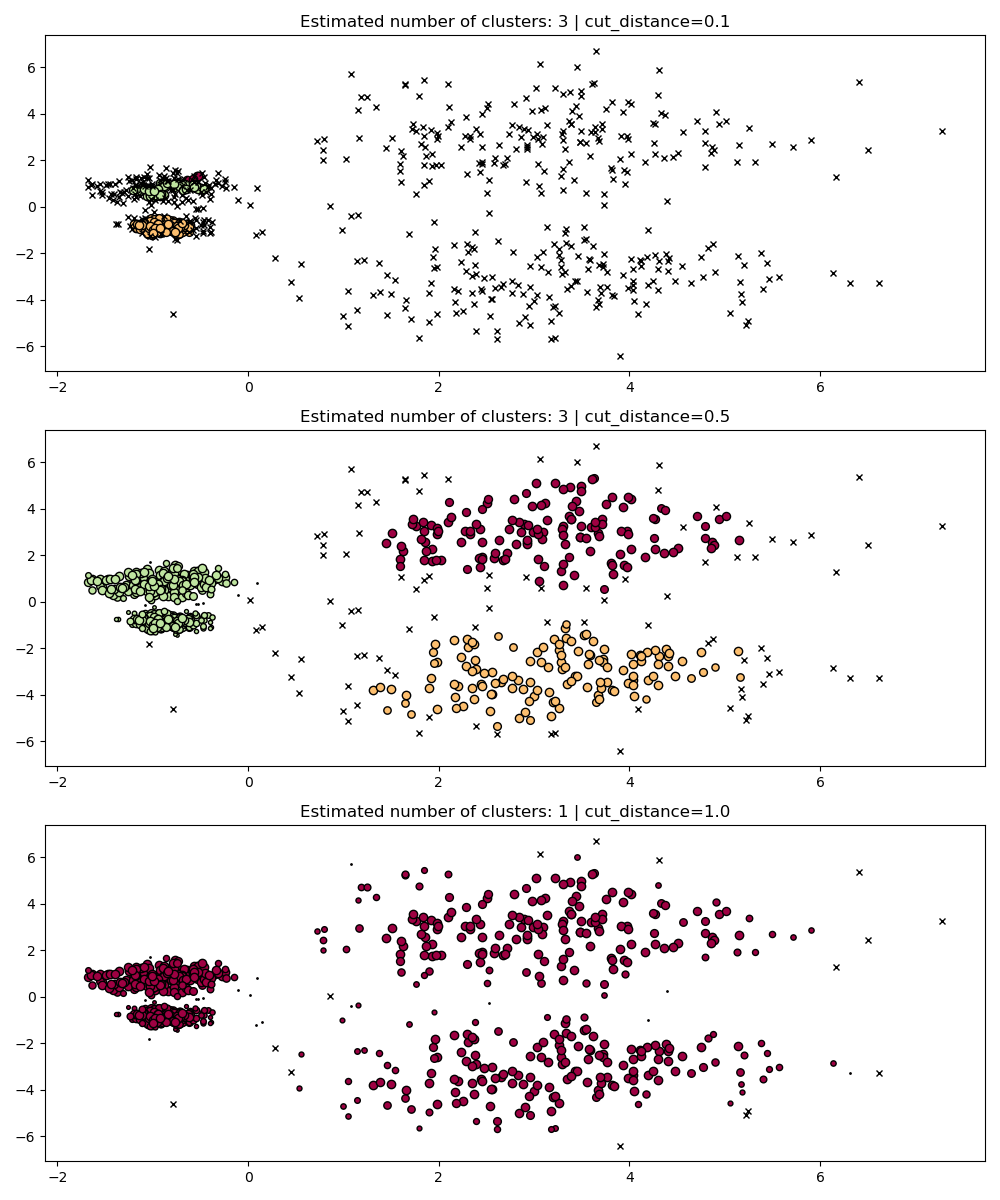
dbscan_clustering#
在 fit 過程中,HDBSCAN 會建立一個單連結樹,該樹編碼了所有點在 DBSCAN 的 eps 參數的所有值上的叢集分群結果。因此,我們可以有效率地繪製和評估這些叢集分群,而無需完全重新計算諸如核心距離、相互可達性和最小生成樹等中間值。我們只需要指定想要進行叢集分群的 cut_distance (等同於 eps) 即可。
PARAM = (
{"cut_distance": 0.1},
{"cut_distance": 0.5},
{"cut_distance": 1.0},
)
hdb = HDBSCAN()
hdb.fit(X)
fig, axes = plt.subplots(len(PARAM), 1, figsize=(10, 12))
for i, param in enumerate(PARAM):
labels = hdb.dbscan_clustering(**param)
plot(X, labels, hdb.probabilities_, param, ax=axes[i])
腳本總執行時間: (0 分鐘 15.245 秒)
相關範例