注意
跳至結尾下載完整的範例程式碼。或透過 JupyterLite 或 Binder 在您的瀏覽器中執行此範例
使用多項式核心近似的可擴展學習#
此範例說明如何使用 PolynomialCountSketch 有效生成多項式核心特徵空間近似。這用於訓練線性分類器,以近似核心化分類器的準確性。
我們使用 Covtype 數據集 [2],嘗試重現 Tensor Sketch [1] 原始論文中的實驗,即 PolynomialCountSketch 實作的演算法。
首先,我們計算原始特徵上線性分類器的準確性。然後,我們在由 PolynomialCountSketch 生成的不同數量特徵(n_components)上訓練線性分類器,以可擴展的方式近似核心化分類器的準確性。
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
準備數據#
載入 Covtype 數據集,其中包含 581,012 個樣本,每個樣本有 54 個特徵,分佈在 6 個類別中。此數據集的目標是僅從製圖變數(沒有遙感數據)預測森林覆蓋類型。載入後,我們將其轉換為二元分類問題,以匹配 LIBSVM 網頁 [2] 中數據集的版本,這是 [1] 中使用的版本。
from sklearn.datasets import fetch_covtype
X, y = fetch_covtype(return_X_y=True)
y[y != 2] = 0
y[y == 2] = 1 # We will try to separate class 2 from the other 6 classes.
分割數據#
這裡我們選擇 5,000 個樣本進行訓練,10,000 個樣本進行測試。要實際重現原始 Tensor Sketch 論文中的結果,請選擇 100,000 個進行訓練。
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(
X, y, train_size=5_000, test_size=10_000, random_state=42
)
特徵正規化#
現在將特徵縮放到 [0, 1] 範圍以匹配 LIBSVM 網頁中數據集的格式,然後如原始 Tensor Sketch 論文 [1] 中所做的那樣正規化為單位長度。
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import MinMaxScaler, Normalizer
mm = make_pipeline(MinMaxScaler(), Normalizer())
X_train = mm.fit_transform(X_train)
X_test = mm.transform(X_test)
建立基準模型#
作為基準,在原始特徵上訓練線性 SVM 並印出準確性。我們也測量並儲存準確性和訓練時間,以便稍後繪製它們。
import time
from sklearn.svm import LinearSVC
results = {}
lsvm = LinearSVC()
start = time.time()
lsvm.fit(X_train, y_train)
lsvm_time = time.time() - start
lsvm_score = 100 * lsvm.score(X_test, y_test)
results["LSVM"] = {"time": lsvm_time, "score": lsvm_score}
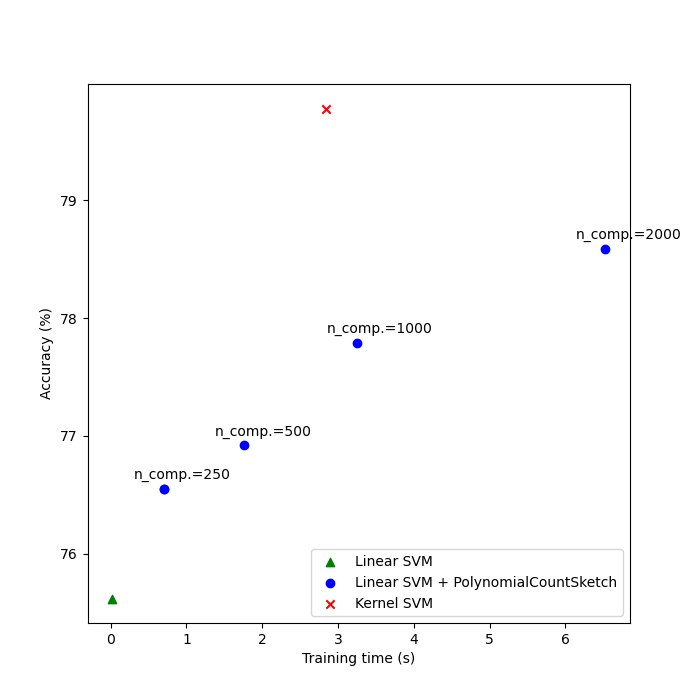
print(f"Linear SVM score on raw features: {lsvm_score:.2f}%")
Linear SVM score on raw features: 75.62%
建立核心近似模型#
然後,我們在由 PolynomialCountSketch 生成的特徵上訓練線性 SVM,並為 n_components 使用不同的值,顯示這些核心特徵近似提高了線性分類的準確性。在典型的應用場景中,n_components 應該大於輸入表示中的特徵數量,才能相對於線性分類實現改進。根據經驗,評估分數/運行時間成本的最佳值通常在 n_components = 10 * n_features 左右實現,儘管這可能取決於正在處理的特定數據集。請注意,由於原始樣本有 54 個特徵,因此四次多項式核心的顯式特徵映射大約有 850 萬個特徵(準確地說,54^4)。感謝 PolynomialCountSketch,我們可以將該特徵空間的大部分判別資訊濃縮到更緊湊的表示中。雖然我們在這個範例中只執行一次實驗(n_runs = 1),但在實務上,應該重複實驗幾次,以補償 PolynomialCountSketch 的隨機性質。
from sklearn.kernel_approximation import PolynomialCountSketch
n_runs = 1
N_COMPONENTS = [250, 500, 1000, 2000]
for n_components in N_COMPONENTS:
ps_lsvm_time = 0
ps_lsvm_score = 0
for _ in range(n_runs):
pipeline = make_pipeline(
PolynomialCountSketch(n_components=n_components, degree=4),
LinearSVC(),
)
start = time.time()
pipeline.fit(X_train, y_train)
ps_lsvm_time += time.time() - start
ps_lsvm_score += 100 * pipeline.score(X_test, y_test)
ps_lsvm_time /= n_runs
ps_lsvm_score /= n_runs
results[f"LSVM + PS({n_components})"] = {
"time": ps_lsvm_time,
"score": ps_lsvm_score,
}
print(
f"Linear SVM score on {n_components} PolynomialCountSketch "
+ f"features: {ps_lsvm_score:.2f}%"
)
Linear SVM score on 250 PolynomialCountSketch features: 76.55%
Linear SVM score on 500 PolynomialCountSketch features: 76.92%
Linear SVM score on 1000 PolynomialCountSketch features: 77.79%
Linear SVM score on 2000 PolynomialCountSketch features: 78.59%
建立核心化 SVM 模型#
訓練核心化 SVM 以查看 PolynomialCountSketch 如何近似核心的效能。當然,這可能需要一些時間,因為 SVC 類的擴展性相對較差。這就是核心近似器如此有用的原因
from sklearn.svm import SVC
ksvm = SVC(C=500.0, kernel="poly", degree=4, coef0=0, gamma=1.0)
start = time.time()
ksvm.fit(X_train, y_train)
ksvm_time = time.time() - start
ksvm_score = 100 * ksvm.score(X_test, y_test)
results["KSVM"] = {"time": ksvm_time, "score": ksvm_score}
print(f"Kernel-SVM score on raw features: {ksvm_score:.2f}%")
Kernel-SVM score on raw features: 79.78%
比較結果#
最後,繪製不同方法對其訓練時間的結果。正如我們所見,核心化 SVM 實現了更高的準確性,但其訓練時間要長得多,而且最重要的是,如果訓練樣本的數量增加,其訓練時間將會增長得更快。
import matplotlib.pyplot as plt
fig, ax = plt.subplots(figsize=(7, 7))
ax.scatter(
[
results["LSVM"]["time"],
],
[
results["LSVM"]["score"],
],
label="Linear SVM",
c="green",
marker="^",
)
ax.scatter(
[
results["LSVM + PS(250)"]["time"],
],
[
results["LSVM + PS(250)"]["score"],
],
label="Linear SVM + PolynomialCountSketch",
c="blue",
)
for n_components in N_COMPONENTS:
ax.scatter(
[
results[f"LSVM + PS({n_components})"]["time"],
],
[
results[f"LSVM + PS({n_components})"]["score"],
],
c="blue",
)
ax.annotate(
f"n_comp.={n_components}",
(
results[f"LSVM + PS({n_components})"]["time"],
results[f"LSVM + PS({n_components})"]["score"],
),
xytext=(-30, 10),
textcoords="offset pixels",
)
ax.scatter(
[
results["KSVM"]["time"],
],
[
results["KSVM"]["score"],
],
label="Kernel SVM",
c="red",
marker="x",
)
ax.set_xlabel("Training time (s)")
ax.set_ylabel("Accuracy (%)")
ax.legend()
plt.show()
參考文獻#
[1] Pham, Ninh 和 Rasmus Pagh。“透過顯式特徵映射的快速且可擴展的多項式核心。”KDD ‘13 (2013)。 https://doi.org/10.1145/2487575.2487591
[2] LIBSVM 二元數據集儲存庫 https://www.csie.ntu.edu.tw/~cjlin/libsvmtools/datasets/binary.html
腳本的總運行時間: (0 分鐘 23.161 秒)
相關範例