注意
跳到結尾下載完整的範例程式碼。或透過 JupyterLite 或 Binder 在您的瀏覽器中執行此範例
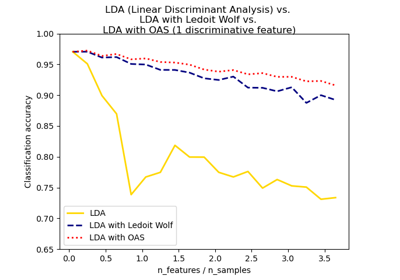
具有共變異數橢圓的線性與二次判別分析#
此範例繪製每個類別的共變異數橢圓,以及 LinearDiscriminantAnalysis (LDA) 和 QuadraticDiscriminantAnalysis (QDA) 學習到的決策邊界。橢圓顯示每個類別的雙標準差。使用 LDA 時,所有類別的標準差都相同,而使用 QDA 時,每個類別都有自己的標準差。
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
資料產生#
首先,我們定義一個函數來產生合成資料。它會建立兩個以 (0, 0) 和 (1, 1) 為中心的 Blob。每個 Blob 都會被指派一個特定的類別。Blob 的分散程度由參數 cov_class_1 和 cov_class_2 控制,這是在從高斯分佈產生樣本時使用的共變異數矩陣。
import numpy as np
def make_data(n_samples, n_features, cov_class_1, cov_class_2, seed=0):
rng = np.random.RandomState(seed)
X = np.concatenate(
[
rng.randn(n_samples, n_features) @ cov_class_1,
rng.randn(n_samples, n_features) @ cov_class_2 + np.array([1, 1]),
]
)
y = np.concatenate([np.zeros(n_samples), np.ones(n_samples)])
return X, y
我們產生三個資料集。在第一個資料集中,兩個類別共享相同的共變異數矩陣,而此共變異數矩陣的特性是球形的 (等向的)。第二個資料集與第一個資料集相似,但並未強制共變異數為球形的。最後,第三個資料集每個類別都有非球形的共變異數矩陣。
covariance = np.array([[1, 0], [0, 1]])
X_isotropic_covariance, y_isotropic_covariance = make_data(
n_samples=1_000,
n_features=2,
cov_class_1=covariance,
cov_class_2=covariance,
seed=0,
)
covariance = np.array([[0.0, -0.23], [0.83, 0.23]])
X_shared_covariance, y_shared_covariance = make_data(
n_samples=300,
n_features=2,
cov_class_1=covariance,
cov_class_2=covariance,
seed=0,
)
cov_class_1 = np.array([[0.0, -1.0], [2.5, 0.7]]) * 2.0
cov_class_2 = cov_class_1.T
X_different_covariance, y_different_covariance = make_data(
n_samples=300,
n_features=2,
cov_class_1=cov_class_1,
cov_class_2=cov_class_2,
seed=0,
)
繪圖函數#
以下程式碼用於繪製所使用估計器的數個資訊片段,也就是 LinearDiscriminantAnalysis (LDA) 和 QuadraticDiscriminantAnalysis (QDA)。顯示的資訊包括
基於估計器的機率估計值的決策邊界;
具有圓圈的散佈圖,代表分類正確的樣本;
具有交叉符號的散佈圖,代表分類錯誤的樣本;
每個類別的平均值,由估計器估計,並以星號標記;
以平均值 2 個標準差的橢圓表示的估計共變異數。
import matplotlib as mpl
from matplotlib import colors
from sklearn.inspection import DecisionBoundaryDisplay
def plot_ellipse(mean, cov, color, ax):
v, w = np.linalg.eigh(cov)
u = w[0] / np.linalg.norm(w[0])
angle = np.arctan(u[1] / u[0])
angle = 180 * angle / np.pi # convert to degrees
# filled Gaussian at 2 standard deviation
ell = mpl.patches.Ellipse(
mean,
2 * v[0] ** 0.5,
2 * v[1] ** 0.5,
angle=180 + angle,
facecolor=color,
edgecolor="black",
linewidth=2,
)
ell.set_clip_box(ax.bbox)
ell.set_alpha(0.4)
ax.add_artist(ell)
def plot_result(estimator, X, y, ax):
cmap = colors.ListedColormap(["tab:red", "tab:blue"])
DecisionBoundaryDisplay.from_estimator(
estimator,
X,
response_method="predict_proba",
plot_method="pcolormesh",
ax=ax,
cmap="RdBu",
alpha=0.3,
)
DecisionBoundaryDisplay.from_estimator(
estimator,
X,
response_method="predict_proba",
plot_method="contour",
ax=ax,
alpha=1.0,
levels=[0.5],
)
y_pred = estimator.predict(X)
X_right, y_right = X[y == y_pred], y[y == y_pred]
X_wrong, y_wrong = X[y != y_pred], y[y != y_pred]
ax.scatter(X_right[:, 0], X_right[:, 1], c=y_right, s=20, cmap=cmap, alpha=0.5)
ax.scatter(
X_wrong[:, 0],
X_wrong[:, 1],
c=y_wrong,
s=30,
cmap=cmap,
alpha=0.9,
marker="x",
)
ax.scatter(
estimator.means_[:, 0],
estimator.means_[:, 1],
c="yellow",
s=200,
marker="*",
edgecolor="black",
)
if isinstance(estimator, LinearDiscriminantAnalysis):
covariance = [estimator.covariance_] * 2
else:
covariance = estimator.covariance_
plot_ellipse(estimator.means_[0], covariance[0], "tab:red", ax)
plot_ellipse(estimator.means_[1], covariance[1], "tab:blue", ax)
ax.set_box_aspect(1)
ax.spines["top"].set_visible(False)
ax.spines["bottom"].set_visible(False)
ax.spines["left"].set_visible(False)
ax.spines["right"].set_visible(False)
ax.set(xticks=[], yticks=[])
LDA 與 QDA 的比較#
我們比較所有三個資料集上的兩個估計器 LDA 和 QDA。
import matplotlib.pyplot as plt
from sklearn.discriminant_analysis import (
LinearDiscriminantAnalysis,
QuadraticDiscriminantAnalysis,
)
fig, axs = plt.subplots(nrows=3, ncols=2, sharex="row", sharey="row", figsize=(8, 12))
lda = LinearDiscriminantAnalysis(solver="svd", store_covariance=True)
qda = QuadraticDiscriminantAnalysis(store_covariance=True)
for ax_row, X, y in zip(
axs,
(X_isotropic_covariance, X_shared_covariance, X_different_covariance),
(y_isotropic_covariance, y_shared_covariance, y_different_covariance),
):
lda.fit(X, y)
plot_result(lda, X, y, ax_row[0])
qda.fit(X, y)
plot_result(qda, X, y, ax_row[1])
axs[0, 0].set_title("Linear Discriminant Analysis")
axs[0, 0].set_ylabel("Data with fixed and spherical covariance")
axs[1, 0].set_ylabel("Data with fixed covariance")
axs[0, 1].set_title("Quadratic Discriminant Analysis")
axs[2, 0].set_ylabel("Data with varying covariances")
fig.suptitle(
"Linear Discriminant Analysis vs Quadratic Discriminant Analysis",
y=0.94,
fontsize=15,
)
plt.show()
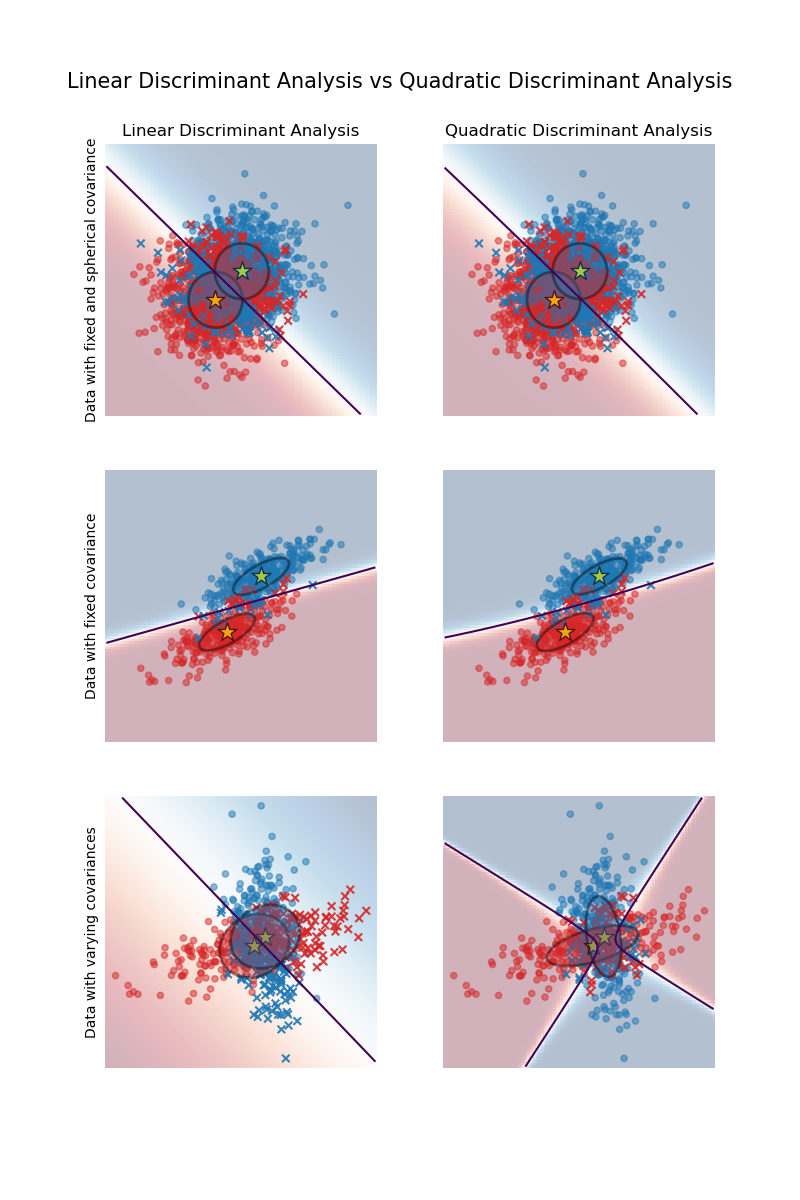
首先要注意的重要事項是,LDA 和 QDA 對於第一個和第二個資料集是等效的。實際上,主要差異在於 LDA 假設每個類別的共變異數矩陣相等,而 QDA 會估計每個類別的共變異數矩陣。由於在這些情況下,資料產生過程對於兩個類別具有相同的共變異數矩陣,因此 QDA 會估計兩個 (幾乎) 相等的共變異數矩陣,因此等效於 LDA 估計的共變異數矩陣。
在第一個資料集中,用於產生資料集的共變異數矩陣是球形的,這會導致判別邊界與兩個平均值之間垂直平分線對齊。對於第二個資料集來說,情況已不再如此。判別邊界僅通過兩個平均值的中間。
最後,在第三個資料集中,我們觀察到 LDA 和 QDA 之間的真正差異。QDA 擬合兩個共變異數矩陣並提供非線性判別邊界,而 LDA 則由於假設兩個類別共享單一共變異數矩陣而擬合不足。
指令碼的總執行時間: (0 分鐘 0.409 秒)
相關範例